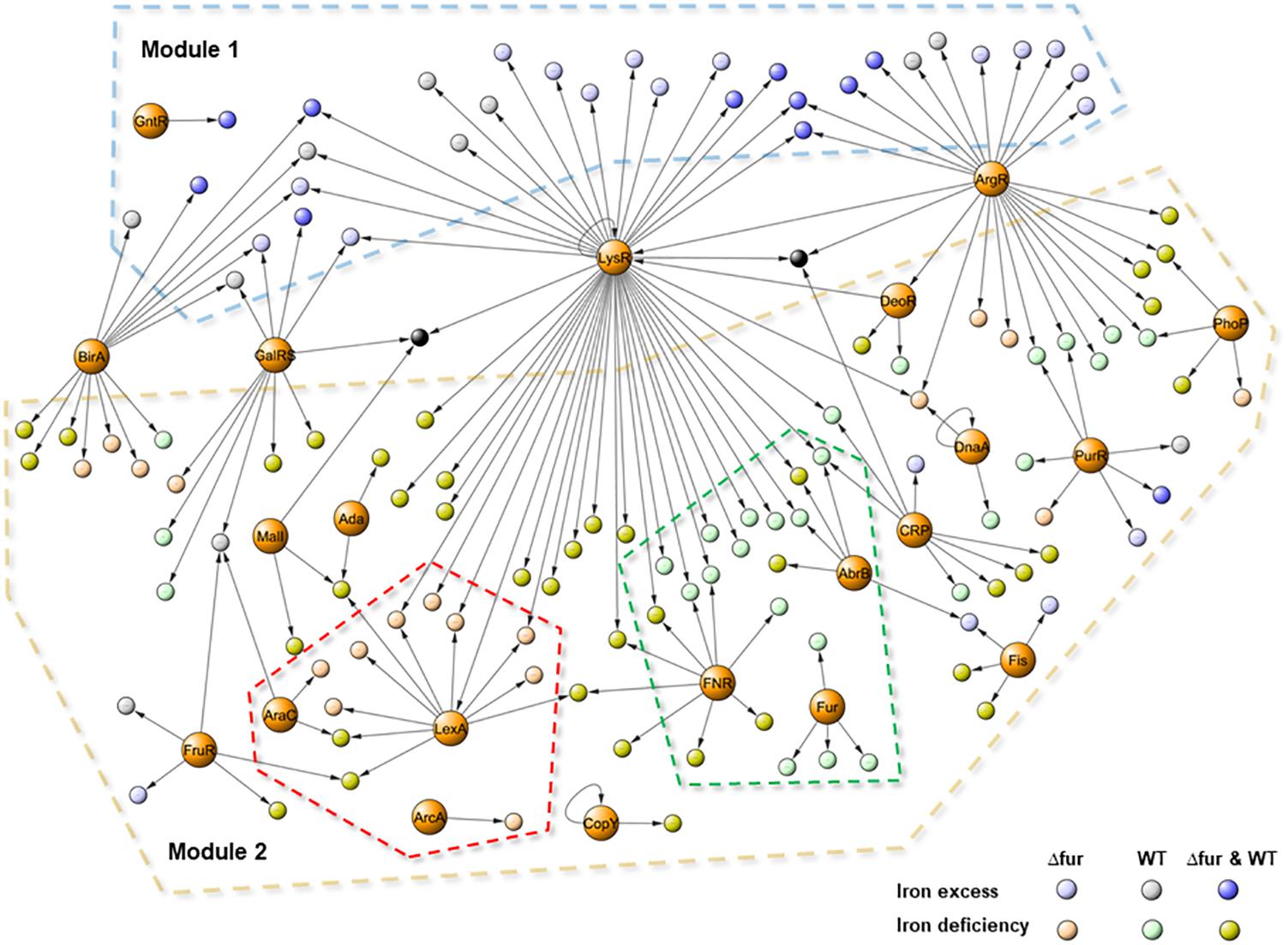
Actividad transcripcional y homeostasis del hierro en Enterococcus faecalis
La variación de la disponibilidad de hierro en el ambiente provoca que las células bacterianas tengan que implementar un control complejo sobre los mecanismos involucrados en la homeostasis del hierro.
En bacterias, el regulador de consumo del hierro Fur es el principal factor de transcripción que controla la homeostasis del hierro; sin embargo, los detalles sobre esta regulación aún no son completamente claros, por lo que en este trabajo se caracterizó la capacidad de Fur para reconfigurar la red global transcripcional e influir en la homeostasis del hierro en Enterococcus faecalis.
Fur es importante porque no sólo se encarga de controlar el consumo de hierro, también está involucrado en el control de genes que codifican para la síntesis de DNA, metabolismo y formación de biofilms.
El factor transcripcional Fur fue removido de las bacterias, una estrategia que les permitió identificar a sus genes blanco. También determinaron su relevancia sobre la respuesta global transcripcional de E. faecalis en condiciones de deficiencia y de exceso de hierro, demostrando que esta bacteria tiene la capacidad de reconfigurar su red génica, activando otros reguladores transcripcionales que hacen posible el mantenimiento de la homeostasis celular. Con el total de datos obtenidos, lograron identificar los reguladores putativos que controlan la transcripción de estos componentes y hacer una lista de genes diferencialmente expresados para reunirlos gráficamente en un modelo.
El modelo contiene 21 familias reguladoras y 139 operones (330 genes incluidos), que se dividen principalmente en tres tipos de factores transcripcionales:
- Reguladores globales: Involucrados con procesos metabólicos, principalmente con el metabolismo basal (LysR, ArgR, GalRS y BirA).
- Reguladores locales: controlan la expresión de un pequeño número de genes que codifican para proteínas que participan en los mismos procesos que los reguladores globales (Ada, DeoR, DnaA, ArcA, GntR, CopY y Fur).
- Reguladores maestros: Comprenden el resto de la familia de reguladores.
En términos de activación transcripcional, el modelo tuvo dos principales módulos. El primero involucrado con genes activados en condiciones de exceso de hierro y el segundo comprende genes activados en condiciones de déficit de hierro.
De esta manera, se concluye que la ausencia de Fur afecta la concentración interna del hierro y activa los sistemas de consumo de hierro impactando también la concentración interna de otros micronutrientes. El exceso de hierro activa genes y reguladores relacionados con el metabolismo basal. Por último, los efectos de pérdida de control sobre el consumo de hierro, cuando Fur es eliminado, conlleva a que la bacteria reconfigure su regulación transcripcional global.
Referencia:
- Latorre, M., Quenti, D., Travisany, D., Singh, K. V., Murray, B. E., Maass, A., & Cambiazo, V. (2018). The role of Fur in the transcriptional and iron homeostatic response of Enterococcus faecalis. Frontiers in microbiology, 9.