Bacterias asociadas a plantas desérticas y su capacidad para promover la resistencia vegetal al estrés hídrico
Alhagi sparsifolia es una especie de plantas fabáceas que habita en los desiertos y resiste a las condiciones de sequía, por lo que resulta un candidato para la obtención de simbiontes microbianos que favorezcan la resistencia al estrés hídrico. Para explorar su diversidad microbiana se secuenciaron amplicones de la región V3-V4 del gen 16S rRNA. Tras la asignación taxonómica de los amplicones, se encontró (al igual que en otras especies de plantas) una mayor diversidad en la rizósfera que en la endósfera de raíz, en la cual predominan las Gammaproteobacterias. El incremento en la abundancia relativa del orden anterior, está acompañada de una reducción de las Alphaproteobacterias, lo cual sugiere que para A. sparsifolia, las Gammaproteobacterias son mejores colonizadoras de la endósfera.

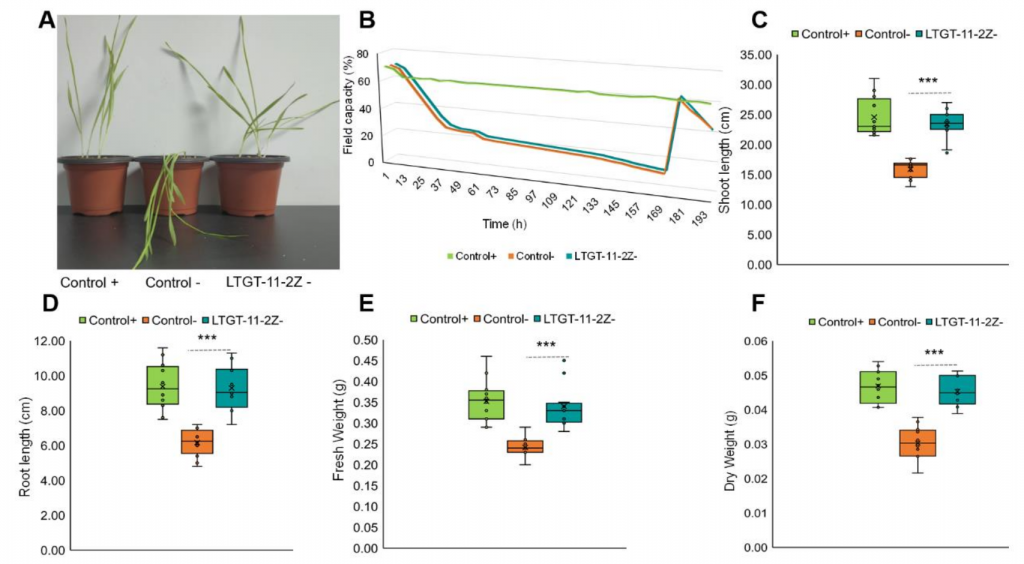
Mientras que en la rizósfera, domina el género Kocuria, Halomonas, Pseudomonas, Truepera y Planoccocus; en la endósfera se encontraron bacterias de los géneros Pseudomonas, Stenotrophomonas y Achromobacter. Con base en los análisis independientes de cultivo, se aislaron siete cepas para probar su efecto en la resistencia de las plantas a la sequía. De las cepas analizadas, se observó que Pseudomonas sp. LTGT-11-2Z tenía un efecto positivo en el crecimiento de las plantas expuestas al estrés hídrico, en las que favoreció el crecimiento de la raíz, la parte aérea y provocó efectos positivos en el peso seco y húmedo de las plantas.
Una vez detectada la cepa promotora del crecimiento vegetal, se secuenció y ensambló su genoma. Dentro de los genes codificados por el genoma de Pseudomonas sp. LTGT-11-2Z, se encontraron glucosidasas, las cuales podrían estar implicadas en la penetración de la pared celular vegetal y la posterior colonización; así como genes implicados en la síntesis de ACC deaminasas, sideróforos y espermidina, rasgos relacionados con la promoción del crecimiento vegetal. Posteriormente se realizó un análisis comparativo con otras bacterias endófitas promotoras del crecimiento vegetal (caracterizadas en estudios previos). De acuerdo con la anotación en KEGG, se encontraron 1,760 genes compartidos entre los cinco genomas, de los cuales 63 fueron exclusivos para Pseudomonas sp. LTGT-11-2Z, los cuales incluyen genes reacionados con el sistema de secreción T6SS y la formación de biopelícula, lo que sugiere que podría haber mecanismos únicos mediando la interacción entre algunas bacterias y plantas.

Auque el estudio está restringido a una sola especie de planta desértica y se analiza la inoculación de cepas de manera individual en un ambiente controlado, los resultados del trabajo muestran que los microorganismos capaces de interactuar con las plantas en condiciones de estrés hídrico, podrían ser un importante reservorio de microorganismos capaces de favorecer el crecimiento de las plantas bajo condiciones de estrés hídrico.
Referencia:
Zhang, L., Zhang, W., Li, Q., Cui, R., Wang, Z., Wang, Y., … & Shen, X. (2020). Deciphering the root endosphere microbiome of the desert plant Alhagi sparsifolia for drought resistance-promoting bacteria. Applied and Environmental Microbiology.




