Influencia del suelo, prácticas agrícolas y genotipo del hospedero en la estructuración de la microbiota de raíz
En éste trabajo se evaluó el efecto de ocho suelos distintos (colectados en África y Europa), ocho genotipos de trigo y dos tipos de manejo agrícola, para evaluar los factores más relevantes en la estructuración de la microbiota de raíz. Para ello se empleó la secuenciación masiva del gen 16S rRNA y de la región ITS, con la finalidad de evaluar la diversidad de bacterias y de eucariontes respectivamente.
En las primeras partes del análisis, evaluaron la relación entre el genotipo del trigo y la diversidad de microorganismos de la raíz, encontrando que aunque la diversidad alfa variabla entre los distintos genotipos, los análisis de diversidad beta llevados a cabo mediante ordenamientos, no mostraron diferencias de acuerdo con el genotipo del hospedero. Sin embargo, cuando se analizó la microbiota en función del suelo, encontraron diferencias a nivel de diversidad alfa, en las que cabe resaltar, la diversidad de procariontes mostró una correlación positiva con la diversidad de procariontes. Además, los análisis de diversidad beta mostraron diferencias entre los suelos africanos y los europeos, lo cual sugiere que en el caso del trigo y al menos a escala continental, el suelo es un mayor predictor de la estruración de la microbiota de raíz, de lo que lo es el genotipo.

Cuando se evaluó la infuencia del manejo agrícola, en localidades ubicadas en Francia e Italia, se encontraron diferencias estadísticamente significativas en la diversidad alfa en los suelos de Italia, dónde el manejo orgánico mostró mayor diversidad. Sin embargo, al evaluar la diversidad beta, se encontraron diferencias relacionadas al manejo agrícola y al sitio de colecta. Las diferencias encontradas están ligadas con el enriquecimiento de 51 taxa en el manejo tradicional y 17 en el manejo agrícola. De los taxa enriquecidos en el manejo agrícola, la mitad correspondieron a eucariotes (Cercozoa, Fungi y Oomycetes) y la mitad restante a bacterias, por lo cual los autores señalan la importancia de los eucariontes en la microbiota.

Considerando la totalidad de los suelos evaluados, las bacterias más diversas correspondieron a Burkholderiaceae, Chithinophagaceae y Flavobacteriaceae; mientras que las comunidades de eucariontes estuvieron dominadas por Sordariomycetes, Peronosporales y Chytridiomycetes. Al tratar de definir un grupo de microorganismos relevantes específicos para cada región, identificaron un microbioma núcleo para Africa, otro para Europa y los comúnes para ambos contienentes. El grupo compartido entre ambos consistió en 2 arqueas, 103 bacterias, 41 hongos y 31 protistas, que aunque comprenden solamente el 2.8% de la diversidad total, tienen una abundancia relativa de 50.2%. Estos microorganismos incluyen al hongo sapótrofo Mortierella; así como la bacterias Massilia y Bradyrhizobium japopinus, la primera capaz de colonizar la raíz y la segunda caracterizada como fijadora de nitrógeno.
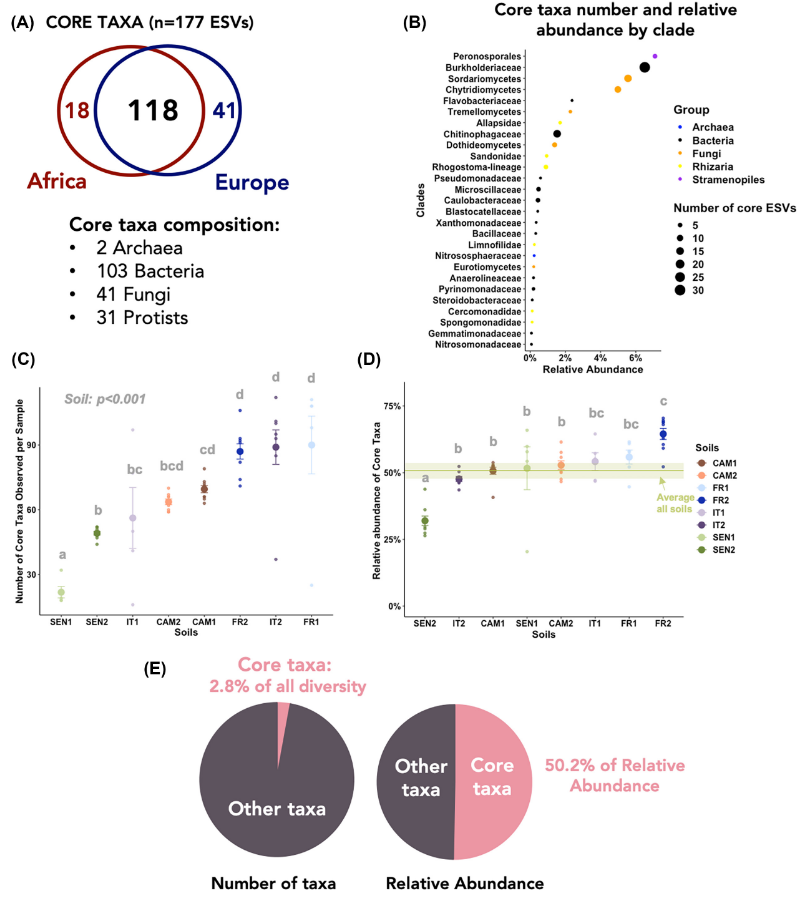
Este trabajo ayuda a entender los principales factores que determinan la microbiota asociada a raíz de trigo, sin embargo, para lograr una mayor comprensión del sistema de estudio, se requieren otro tipo de estudios que podrían incluir metagenómica, cultivo y creación de microbiomas sintéticos.
Referencia:
Simonin, M., Dasilva, C., Terzi, V., Ngonkeu, E. L., Diouf, D., Kane, A., … & Moulin, L. (2020). Influence of plant genotype and soil on the wheat rhizosphere microbiome: Evidences for a core microbiome across eight African and European soils. FEMS Microbiology Ecology, 96(6), fiaa067.



