Expandiendo la diversidad de virus en ambientes extremos
En los últimos años, los avances tecnológicos han permitido estudiar virus en ambientes extremos que antes parecían inaccesibles. Tradicionalmente, para investigar un virus era necesario cultivar a su hospedador en el laboratorio, lo que limitaba mucho el alcance de los estudios, ya que muchos microorganismos de ambientes extremos no pueden crecer fácilmente en condiciones de laboratorio. Sin embargo, la metagenómica ha revolucionado el campo de la virología, permitiendo analizar directamente el material genético de virus presentes en muestras ambientales sin necesidad de cultivar a sus hospedadores.
Las muestras analizadas en este trabajo provienen de una gran variedad de ambientes extremos alrededor del mundo. Entre los sitios de muestreo se destacan las fuentes termales del Parque Nacional de Yellowstone en Estados Unidos, respiraderos hidrotermales en el fondo marino, manantiales volcánicos de Kamchatka en Rusia y otros puntos de difícil acceso. Estos lugares se caracterizan por condiciones extremas de temperatura, acidez o salinidad, y han sido explorados tanto mediante estudios metagenómicos como por investigaciones específicas de virus aislados.
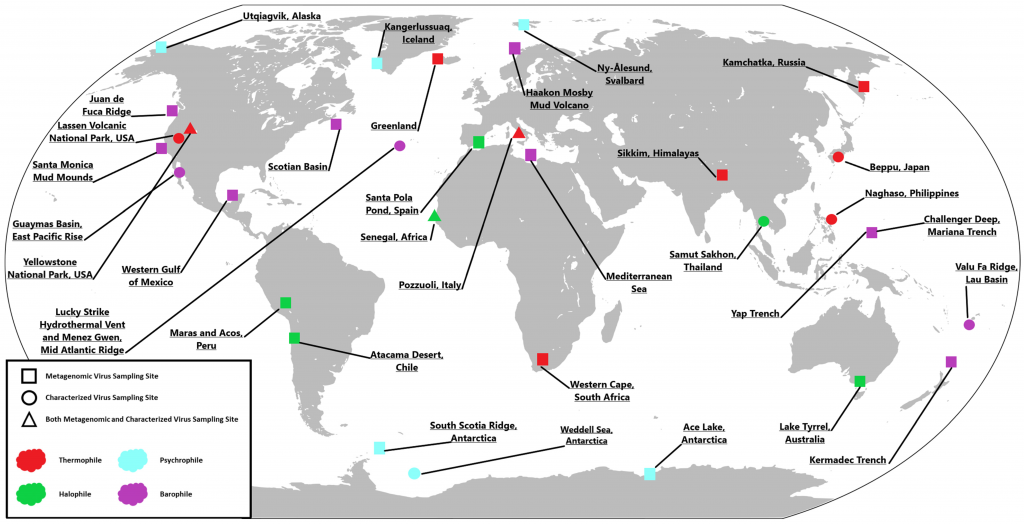
Figure 1. Global Sampling Map. Sampling locations from around the globe featured in this review. Both metagenomic and specific viral studies were conducted utilizing samples taken from the above locations. World map obtained from [18].
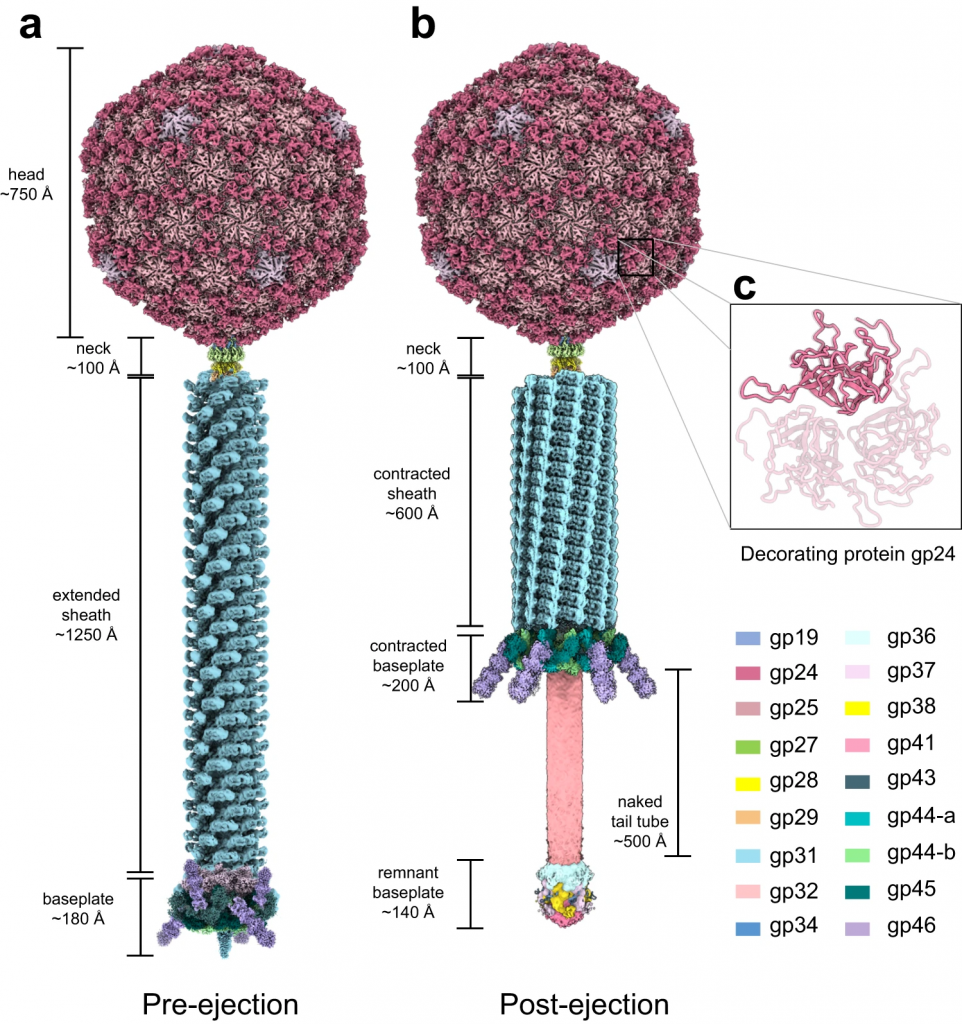
Uno de los hallazgos más novedosos es la descripción del bacteriófago P74-26, también llamado el “virus Rapunzel” por la longitud de su cola, que infecta bacterias termofílicas del género Thermus, virus aislado de aguas termales en el Parque Nacional de Yellowstone, que se ha convertido en un modelo para estudiar cómo los virus pueden adaptarse y sobrevivir en condiciones de calor extremo, la cápside del virus está rodeada por una proteína decoradora llamada gp87, que forma una especie de jaula molecular alrededor de toda la cápside. Esta jaula está formada por trímeros de gp87 que se entrelazan con las proteínas de la cápside a través de interacciones tipo “lazo”, creando una red de enlaces que refuerzan la estabilidad del virus. Este diseño no solo protege al virus de la desnaturalización por el calor, sino que también le permite expandirse durante el empaquetamiento del genoma, manteniendo la integridad estructural incluso bajo condiciones extremas.
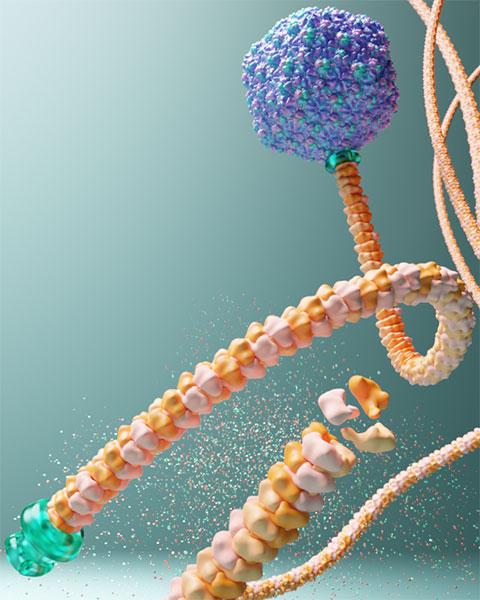
Courtesy of Leonora Martinez-Nunez
Compared with most phages, P74-26 uses half the number of building blocks to form stacking rings that make up the tail.

The bacteriophage P74-26 has a tail 10 times longer than most other phage tails and is nearly 1 micrometer long, about the width of some spider’s silk.
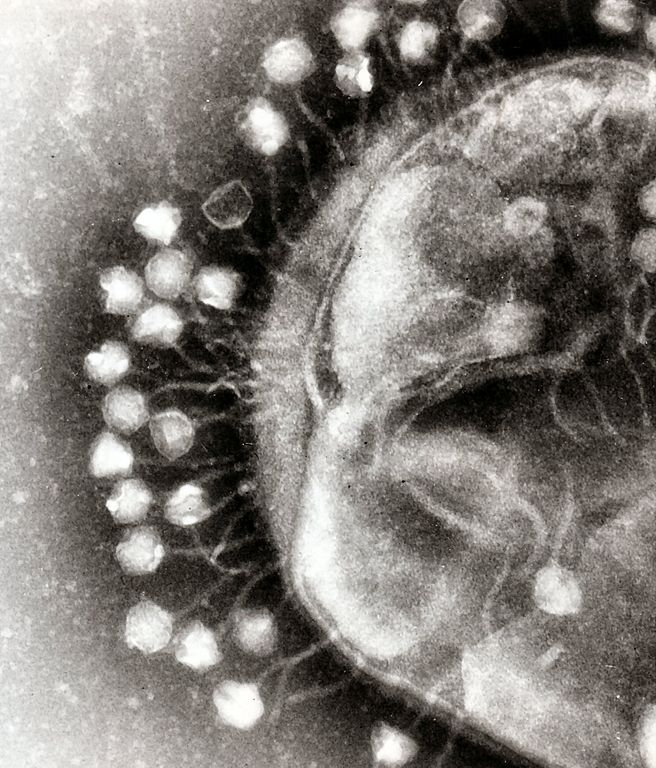
Professor Graham Beards on Wikimedia Commons
Transmission electron micrograph of multiple bacteriophages attached to a bacterial cell wall; the magnification is approximately 200,000. Figuras tomadas de Marissa Locke Rottinghaus. (2023). The ‘Rapunzel’ virus: an evolutionary oddity. https://www.asbmb.org/asbmb-today/science/031423/the-rapunzel-virus
La proteína gp87 es especialmente interesante porque posee un dominio β-tulipán, similar al de ciertas proteínas anti-CRISPR, lo que sugiere una posible relación evolutiva. Además, los residuos hidrofóbicos en el núcleo de gp87 contribuyen a su extraordinaria estabilidad, una característica crucial para sobrevivir en ambientes termales. Esta arquitectura única convierte a P74-26 en uno de los virus más estables conocidos y es un ejemplo de cómo la evolución puede generar soluciones estructurales innovadoras para enfrentar desafíos ambientales extremos.
Composite cryo-EM reconstructions of the E217 virion at neutral pH (a) and after incubation at alkaline pH (b). c Zoom-in view of the trimeric cementing protein gp25 that decorates the icosahedral capsid. Figura tomada de Li, F., Hou, C.-F. D., Lokareddy, R. K., Yang, R., Forti, F., Briani, F., & Cingolani, G. (2023). High-resolution cryo-EM structure of the Pseudomonas bacteriophage E217. Nature Communications, 14(1), 4052. https://doi.org/10.1038/s41467-023-39756-z
Otro hallazgo llamativo es la variedad de estrategias que estos virus han desarrollado para evadir los sistemas inmunitarios de sus hospedadores. Por ejemplo, los virus que infectan arqueas del género Sulfolobus han evolucionado mecanismos para escapar del sistema CRISPR, que es el principal sistema de defensa de estos microorganismos. En algunos casos, los virus acortan o reorganizan su genoma para evitar ser detectados, y en otros introducen mutaciones específicas cerca de las secuencias objetivo del sistema inmune, lo que les permite aumentar su infectividad.
Además, se han observado mecanismos de ensamblaje y liberación viral realmente únicos. Un caso interesante es el del virus Sulfolobus islandicus filamentous virus (SIFV), que induce la formación de pirámides hexagonales en la superficie de la célula hospedadora para facilitar la salida de nuevas partículas virales. Este tipo de estructuras no se había visto antes y sugiere una gran creatividad evolutiva en estos sistemas.
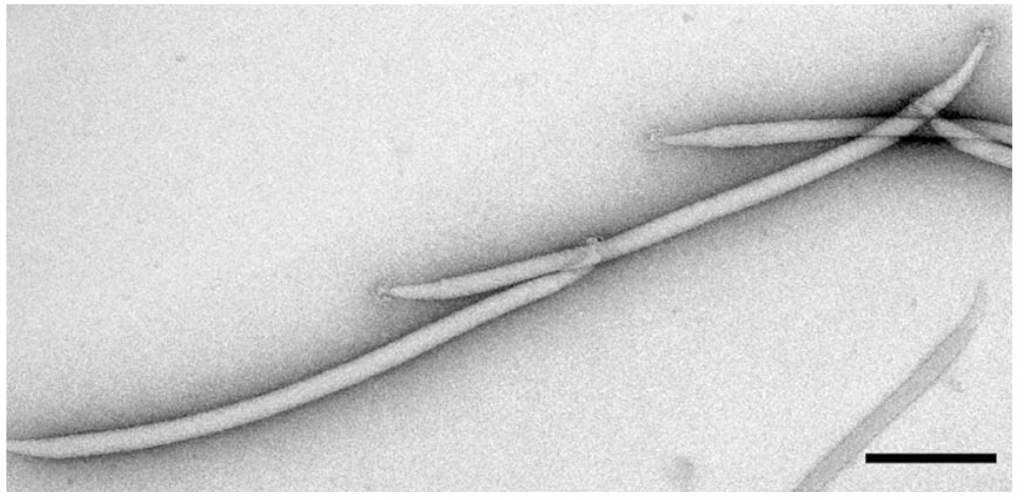
Figure 2. Sulfolobus filamentous virus 1 (SFV1). SFV1 was isolated from samples of Sulfolobus shibatae from the acidic hot springs of Umi Jigoku located in Beppu, Japan. The terminal mop-like structures are also visible. Some viruses were seen with elongated versions of these mop-like structures, reaching up to 700 nm in length. Reprinted from [33]. Scale bar = 200 nm.
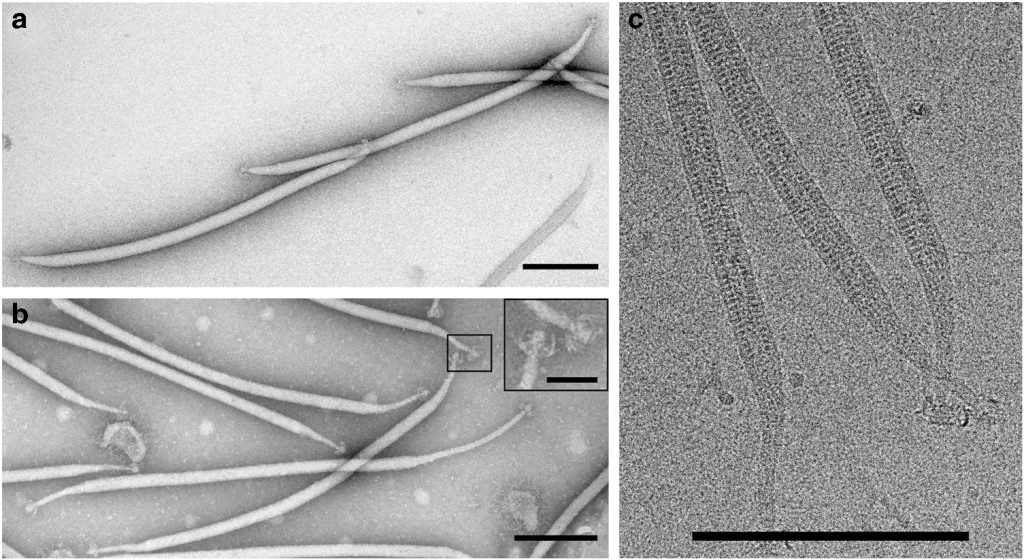
Electron micrographs of SFV1 virions. a Negatively stained purified virions. b Negatively stained virions with an elongated “neck;” mop-like terminal structures are shown in the inset. c Cryo-EM of SFV1. Scale bars, 200 nm (50 nm in the inset, b). Figura tomada de Liu, Y., Osinski, T., Wang, F., Krupovic, M., Schouten, S., Kasson, P., Prangishvili, D., & Egelman, E. H. (2018). Structural conservation in a membrane-enveloped filamentous virus infecting a hyperthermophilic acidophile. Nature Communications, 9(1), 3360. https://doi.org/10.1038/s41467-018-05684-6
Mollivirus sibericum es un virus gigante psicrófilo (adaptado al frío) que ha fascinado a la comunidad científica por su origen y sus características extraordinarias. Fue aislado de una muestra de permafrost de 30,000 años de antigüedad en Chukotka, Rusia, la misma muestra de la que se extrajo el famoso Pithovirus sibericum. Este hallazgo es especialmente relevante porque el permafrost actúa como una especie de “cápsula del tiempo”, conservando organismos y virus antiguos en un estado latente durante milenios. Lo más sorprendente de Mollivirus sibericum es que, a pesar de tener una antigüedad estimada igual a la del permafrost donde se encontró, el virus aún conserva su capacidad de infectar. En experimentos de laboratorio, los investigadores observaron que Mollivirus sibericum puede infectar a la ameba Acanthamoeba castellanii, mostrando actividad viral bajo el microscopio incluso después de 30,000 años de congelamiento. Esto demuestra la increíble resistencia y viabilidad de algunos virus gigantes en condiciones extremas y durante períodos de tiempo asombrosamente largos.
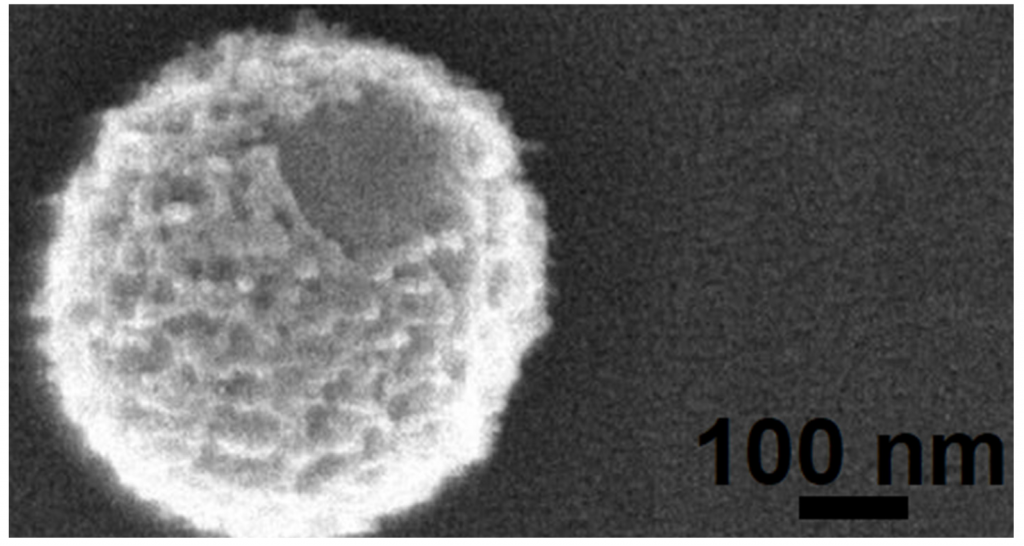
Figure 4. Mollivirus sibericum. This giant psychrophilic virus was isolated from the same 30,000-year-old permafrost sample as Pithovirus sibericum [93] from Chukotka, Russia. The virus is estimated to be as old as the permafrost itself and yet retains its virulence. Infection of the amoeba Acanthamoeba castellanii was seen under microscopy. Reprinted with permission from [94]. Copyright 2015 C. Abergel and J.M. Claverie. Scale bar = 100 nm.
En los últimos años, ha crecido notablemente el interés en las propiedades de los microorganismos y virus que habitan ambientes de bajas temperaturas, conocidos como psicrófilos. Estos organismos, adaptados a condiciones de frío extremo, producen proteínas y enzimas con características únicas que han captado la atención de diversas industrias. Uno de los descubrimientos más valiosos han sido las proteínas de unión al hielo (Ice-Binding Proteins, IBPs), presentes en muchos extremófilos psicrófilos. Estas proteínas tienen la capacidad de interactuar con los cristales de hielo, impidiendo su crecimiento o modificando su estructura. Gracias a esta propiedad, las IBPs pueden ser utilizadas en la industria alimentaria para preservar la calidad de los productos congelados, evitando la formación de grandes cristales de hielo que dañan la textura y el sabor. Además, en el ámbito médico, las IBPs permiten almacenar materiales biológicos, como células y tejidos, a bajas temperaturas sin comprometer su viabilidad, lo cual es fundamental para bancos de sangre, trasplantes y biotecnología.
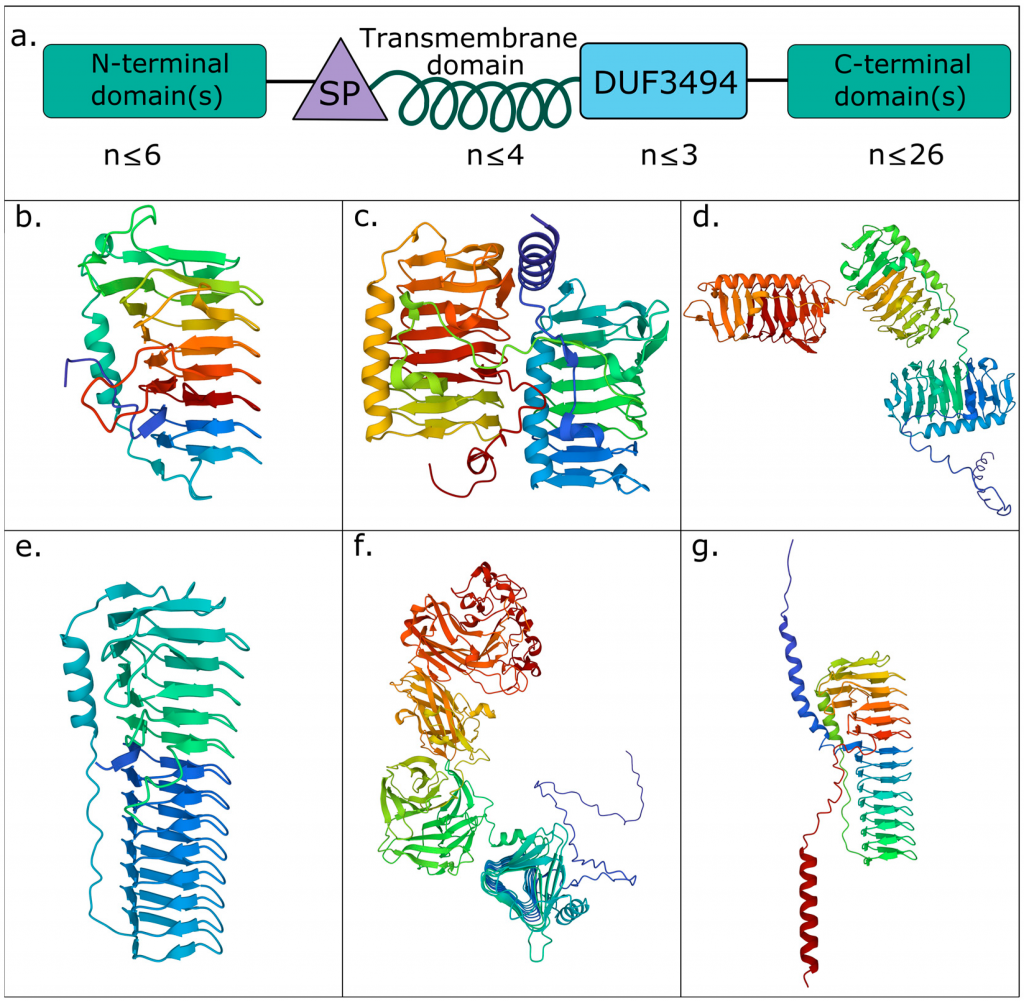
Figure 3. The structures and domain architectures of abundant prokaryotic ice-binding proteins reflect potentially diverse biological roles. (a) Concept diagram displaying the modular diversity of IBP domain architectures. IBPs minimally consist of a single DUF3494 domain (blue) but can consist of up to three DUF3494 domains. Where multiple DUF3494 domains are found, they are not necessarily found in immediate succession—other domains may be interspersed among them. Signal peptides (SP; yellow) and transmembrane domains (TMDs; green helix) are variably present, with up to four transmembrane domains being found in a protein. Additional N and C-terminal domains (pink; location defined by the position of the first DUF3494 domain) are also found, with up to 6 N-terminal domains or up to 26 C-terminal domains in a single protein (up to 28 domains in a single protein). The most abundant IBP domain architectures found in the total assembly were (b) single domain IBPs; (c) double domain IBPs; (d) triple domain IBPs; (e,f) single domain IBPs with an additional C-terminal DUF4842 (pfam16130); (g) single domain IBPs with an additional C-terminal PEP C-term motif (pfam07589). The most environmentally abundant representative of each domain architecture was selected for modelling. Note the length of the DUF3494 domain in (e–g). Proteins are coloured according to residue position, blue being the N-terminus and red being the C-terminus of the protein. Further information about domain architecture abundances is found in Table 1. Figura tomada de Winder, J. C., Boulton, W., Salamov, A., Eggers, S. L., Metfies, K., Moulton, V., & Mock, T. (2023). Genetic and Structural Diversity of Prokaryotic Ice-Binding Proteins from the Central Arctic Ocean. Genes, 14(2), 363. https://doi.org/10.3390/genes14020363
Marinitoga piezophila virus 1, conocido como MPV1, es un virus que representa uno de los ejemplos más fascinantes de la virología de ambientes extremos. Este virus fue aislado de una cepa de Marinitoga piezophila, un microorganismo termófilo que habita en los respiraderos hidrotermales del fondo marino, específicamente en la Dorsal del Pacífico Oriental. Estos respiraderos son ambientes extremos, caracterizados por altas temperaturas, presiones elevadísimas y una química muy particular, lo que convierte a sus habitantes en verdaderos extremófilos. MPV1 pertenece a la familia Siphoviridae, reconocible por su cápside icosaédrica y su larga cola flexible, aunque en este caso se adapta a las condiciones extremas del entorno submarino. Lo que hace a este virus especialmente interesante y novedoso es su capacidad para empaquetar, junto con su propio material genético, un elemento genético móvil de su hospedador: un plásmido de 13.3 kilobases. Es decir, MPV1 no solo transporta su propio ADN, sino que también puede llevar fragmentos de ADN de su huésped bacteriano.
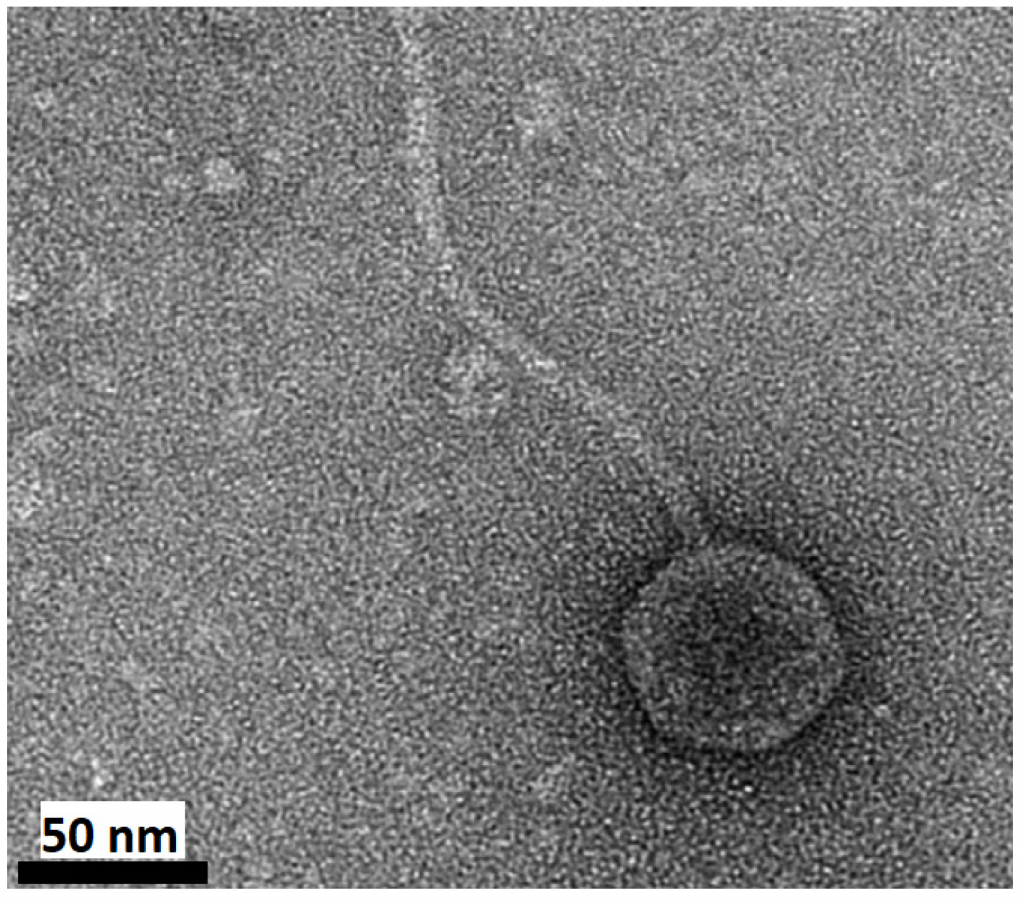
Figure 5. Marinitoga piezophila virus 1 (MPV1). This Siphoviridae-like virus was isolated from a strain of Marinitoga piezophila, a thermophilic microbe found on deep sea hydrothermal vents on the East Pacific Rise. Along with its own genetic material, this virus packages a plasmid-based mobile genetic element from its host, 13.3 kb in size. Reprinted with permission from [21] Copyright 2015 Elsevier. Scale bar = 50 nm.
Todo esto no solo amplía nuestro conocimiento sobre la biodiversidad viral, sino que también abre nuevas posibilidades para aplicaciones biotecnológicas. Por ejemplo, las proteínas virales termostables podrían usarse como vehículos para administrar medicamentos o como herramientas en biotecnología y genética. Además, el estudio de estos virus puede aportar claves sobre la evolución de la vida y la posibilidad de existencia de organismos en ambientes extremos, incluso fuera de la Tierra.
Referencia original:
-Manuel, R. D., & Snyder, J. C. (2024). The Expanding Diversity of Viruses from Extreme Environments. International Journal of Molecular Sciences, 25(6), 3137. https://doi.org/10.3390/ijms25063137
Referencias adicionales de donde fueron tomadas algunas imagenes:
-Virus Rapunzel: Marissa Locke Rottinghaus. (2023). The ‘Rapunzel’ virus: an evolutionary oddity. https://www.asbmb.org/asbmb-today/science/031423/the-rapunzel-virus
-Plegamiento beta-tulipan: Li, F., Hou, C.-F. D., Lokareddy, R. K., Yang, R., Forti, F., Briani, F., & Cingolani, G. (2023). High-resolution cryo-EM structure of the Pseudomonas bacteriophage E217. Nature Communications, 14(1), 4052. https://doi.org/10.1038/s41467-023-39756-z
-Estructura terminal mop-like en virus: Liu, Y., Osinski, T., Wang, F., Krupovic, M., Schouten, S., Kasson, P., Prangishvili, D., & Egelman, E. H. (2018). Structural conservation in a membrane-enveloped filamentous virus infecting a hyperthermophilic acidophile. Nature Communications, 9(1), 3360. https://doi.org/10.1038/s41467-018-05684-6
-Proteinas de unión a hielo: Winder, J. C., Boulton, W., Salamov, A., Eggers, S. L., Metfies, K., Moulton, V., & Mock, T. (2023). Genetic and Structural Diversity of Prokaryotic Ice-Binding Proteins from the Central Arctic Ocean. Genes, 14(2), 363. https://doi.org/10.3390/genes14020363



