Aislamiento de endófitos nuevos del filo Verrucomicrobia
En las plantas terrestres, las raíces son el primer sitio en donde ocurren las interacciones con diversos microorganismos. La colonización de la raíz por parte de bacterias endófitas es un proceso activo que requiere de ciertas características y mecanismos moleculares. Para poder determinar estos mecanismos, se han hecho estudios reduccionistas en donde se han aislado bacterias. Sin embargo, dada la especialización hacia el nicho, son pocas las bacterias endófitas que se han logrado aislar y estudiar. Recientemente, se ha encontrado que el filo Verrucomicrobia se encuentra de manera abundante en la endósfera (hasta el 25 %), pero son muy pocos los estudios que se han hecho sobre este filo. Además, existe un sesgo en las colecciones actuales de bacterias endófitas del filo Verrucomicrobia, por lo que en este estudio se intentó aislar a Verrucomicrobias de rizomas de arroz Oryza sativa y O. longistaminata.
Para aislar a las Verrucomicrobias endófitas, se obtuvo un cultivo mixto de la endósfera y la superficie de la raíz (rizoplano). Después, se hicieron aislados y basados en el patrón de crecimiento, las características de la colonia y la morfología microscópica (cocos) se logró identificar a cuatro posibles Verrucomicrobias. A estas cepas las nombraron: LW23, ER46 y LR76 (estos tres aislados se obtuvieron del rizoplano) y EW11 (aislada de la endosfera). Para determinar las relaciones filogenéticas de los aislados se obtuvo la secuencia de 16s rRNA de las cepas y se llevaron a cabo análisis filogenéticos. Sus resultados muestran que algunas de las cepas aisladas, se agruparon con cepas que no se habían podido cultivar en el laboratorio (Figura 1). Además, dada la ubicación filogenética y la identidad de nucleótidos promedio (ANI) de los cuatro aislados, proponen que la cepa LW23 podría formar una nueva subdivisión dentro del filo Verrucomicrobia.

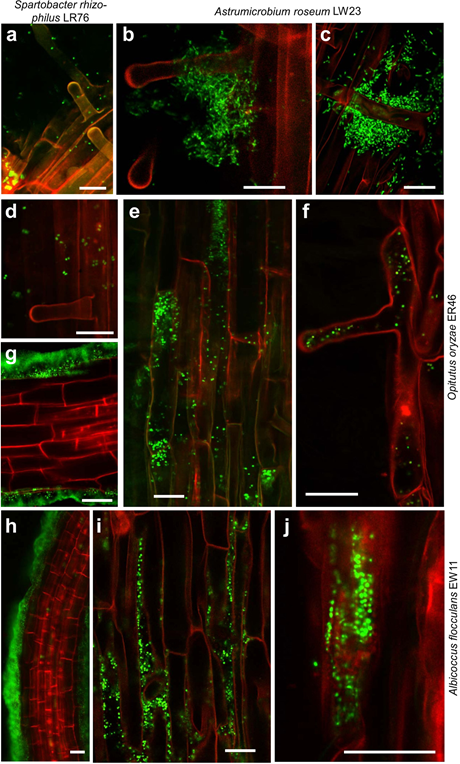
Se ha observado que las bacterias asociadas a la raíz pueden ocupar diferentes nichos: el suelo, la endosfera o el rizoplano, y por lo tanto muestran interacciones y adaptaciones genómicas diferentes que les permiten colonizar estos nichos. Para determinar la capacidad y patrones de colonización de los aislados, se usó un sistema de cultivo hidropónico de plantas de arroz. Los patrones de colonización fueron obtenidos utilizando microscopía con focal laser. La cepa LR76 colonizó el rizoplano, mientras que LW23 se ancló a las raíces y a los pelos radiculares formando agregados. La cepa ER46 colonizó levemente el rizoplano, mientras que la cepa EW11 se observó colonizando el rizoplano y la endosfera, en algunos casos formó biopelículas muy densas. Remarcablemente, en este estudio, se reporta esto como la primera prueba microscópica de colonización endofítica por organismos del filo Verrucomicrobia (Figura 2).
Una vez que se validó que estas Verrucomicrobias aisladas pueden colonizar las raíces de las plantas ya sea como endófitos o colonizando sólo el rizoplano, los investigadores, obtuvieron los genomas en borrador de las cepas que aislaron. Dichos genomas borrador se compararon con otros genomas y metagenomas de bacterias que se sabe habitan la endosfera, el rizoplano y el suelo. Se incluyeron secuencias de los siguientes fila: Proteobacteria, Acidobacteria Gemmatimonadetes, entre otros. La comparación consistió en cuantificar la diversidad de los dominios de las familias de proteínas de los genomas usando la clasificación de Pfam. Los resultados de este análisis mostraron que el número de dominios diferentes fue más alto en los endófitos, seguido por los colonizadores del rizoplano y finalmente por los de suelo. Esto sugiere que existe un incremento en la complejidad de las funciones de las proteínas a medida que incrementa la interacción planta-microorganismo. Así mismo, el análisis de componentes principales de estos dominios confirmó la especialización del nicho, en donde se observa una separación entre los dominios Pfam de las endófitas, rizoplano y las bacterias de suelo. La relación funcional entre los organismos de la endosfera y el rizoplano fue mayor debido a que se observó una mayor intersección de sus dominios Pfam. En los endófitos, observaron un enriquecimiento en dominios que están relacionados a la movilidad por flagelos lo que fue confirmado por ensayos de swarming. Además, el metabolismo de los endófitos parece especializarse hacia las rutas que tienen que ver con el metabolismo de aminoácidos e hidrólisis de carbohidratos. También encontraron genes relacionados con el sistema de dos componentes, el cual parecen tener un papel importante durante la colonización; genes relacionados con la producción de ácido indol acético y fosfatasas (Figura 3). Finalmente, evaluaron el efecto de las 4 cepas de Verrucomicrobia sobre el crecimiento de la raíz de plantas de O. longistaminata, estos experimentos los llevan a cabo utilizando cultivos hidropónicos. Sus resultados muestran que las cepas ER46 (endofítica) y LR76 incrementaron significativamente el peso fresco de la raíz.

Referencia
Bünger, W., Jiang, X., Müller, J., Hurek, T., & Reinhold-Hurek, B. (2020). Novel cultivated endophytic Verrucomicrobia reveal deep-rooting traits of bacteria to associate with plants. Scientific reports, 10(1), 8692.




