Cooperación enzimática entre la cochinilla y su microbiota
La descomposición de la materia orgánica vegetal es un paso muy importante en el ciclo de carbono. Uno de los componentes principales de la biomasa vegetal es la lignocelulosa, que se compone de celulosa, hemicelulosa y lignina, en cuya degradación participan multiples enzimas de las denomidas CAZymes (Carbohydrate-Active enZYmes). En éste proceso participan hongos, bacterias y animales como las termitas, los diplópodos e isópodos terrestres. No obstante, hasta la fecha no se conocen genomas de animales con la capacidad metabólica para transformar polisacáridos vegetales en monómeros de carbohidratos, por lo que para su degradación, requieren de interacciones simbióticas con microorganismos con dichas capacidades. Para estudiar dichas relaciones simbióticas, se utilizaron a organismos de la especie Armadillidium vulgare (cochinillas), en los que se analizaron las transcriptomas del hospedero e información metagenómica de su microbiota. Los organismos analizados fueron obtenidos de linajes crecidos en el laboratorio y en campo, procedentes de diferentes países.
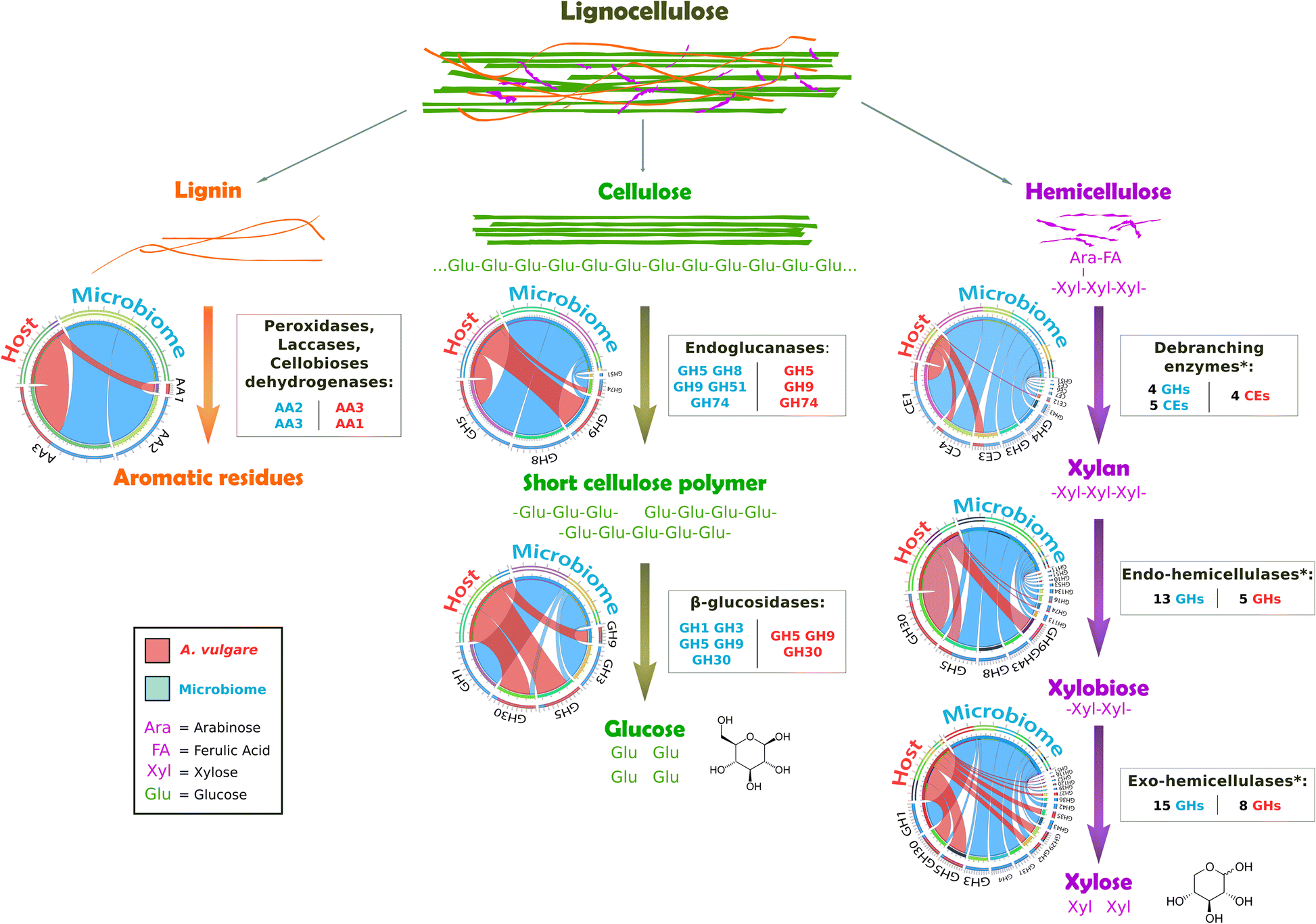
Fig. 1 Model for lignocellulose degradation in the A. vulgare holobiont. Diagrams represent the CAZy families contributed by the host (red) and the microbiome (blue). (I) Lignin would be partially degraded to release cellulose and hemicellulose. (II) Cellulose would be degraded by the action of endoglucanases and β-glucosidases. A high number of β-glucosidases and mechanical fragmentation by A. vulgare could compensate for the lack of exoglucanases. (III) The A. vulgare holobiont could degrade most types of hemicellulose due to the high diversity of Debranching enzymes* (CE1, CE3, CE4, CE5, CE6, CE7, CE12, GH3, GH4, GH43, GH51), Endo-hemicellulases* (GH5, GH8, GH9, GH10, GH11, GH16, GH30, GH43, GH51, GH53, GH74, GH113, GH128, GH134), and Exo-hemicellulases* (GH1, GH2, GH3, GH4, GH5, GH27, GH29, GH30, GH31, GH35, GH36, GH39, GH42, GH43, GH51, GH57, GH116, GH120)
Al comparar la información metatranscriptómica del hospedero, con la meteganómica de la microbiota, fue posible predecir genes que codifican para CAZymes. El conjunto de enzimas de la cochilla y su microbiota, cuentan con la capacidad de degradar la lignina, la celulosa y la hemicelulosa de manera complementaria, aunque la mayor parte de las enzimas detectadas corresponden a la microbiota.

Fig. 4 Relative abundance of prokaryotic taxa contributing lignocellulose-degrading CAZymes depending on a host origin, gender, and Wolbachia infectionstatus, b host origin alone, and c for several genes consistently present in isopods of both field and laboratory origin. See Table 3 for a detailed annotation of these genes
Las hemicelulasas, que son el grupo más abundante detectado de enzimas para la degradación de lignocelulosa, también muestran variaciones en relación con los grupos taxonómicos que las presentan. En el caso de los animales de laboratorio, Kluyvera, Enterobacter y Vibrio, son los géneros que presentan la mayor parte de los genes para la síntesis de hemicelulasas; en el campo, se encuentran en las bacterias Rickettsiella, Wolbachia, Microbacterium y las arqueas Candidatus Nitrosocosmicus y Nitrosphaera.

Fig. 5 Lignocellulose-degrading enzymes and their associated microbial community in (a) isopods from the laboratory and (b) isopods from a natural population. c The microbial taxa contributing lignocellulose-binding modules in both field and laboratory specimens
Como resultado del estudio, se obtuvo evidencia que apoya la colaboración entre A. vulgare y su microbiota en la digestión de la lignocelulosa; al mismo tiempo que se observa redundancia funcional en las comunidades que reclutan los animales en condiciones naturales y en el laboratorio, sin embargo los autores sugieren que se requieren otros estudios para validad estos resultados.
Bredon, M., Dittmer, J., Noël, C., Moumen, B., & Bouchon, D. (2018). Lignocellulose degradation at the holobiont level: teamwork in a keystone soil invertebrate. Microbiome, 6(1), 162.




