Dos genotipos de cebada controlan el reclutamiento de Pseudomonas en sus raíces debido a diferencias en sus exudados radiculares
El reclutamiento de los microorganismos asociados a la raíz es parcialmente determinado por la planta. Las secreción radicular de metabolitos específicos es un mecanismo mediante el cual ocurre la selección de microorganismos durante la colonización de las raíces . En este trabajo se evaluó la composición del microbioma de rizósfera de dos genotipos de cebada, una variedad local domesticada antes de programas modernos de mejor vegetal (Chevallier) y una variedad moderna (Tipple), también se exploró el papel de los exudados radiculares en el reclutamiento de diferentes aislados de Pseudomonas.
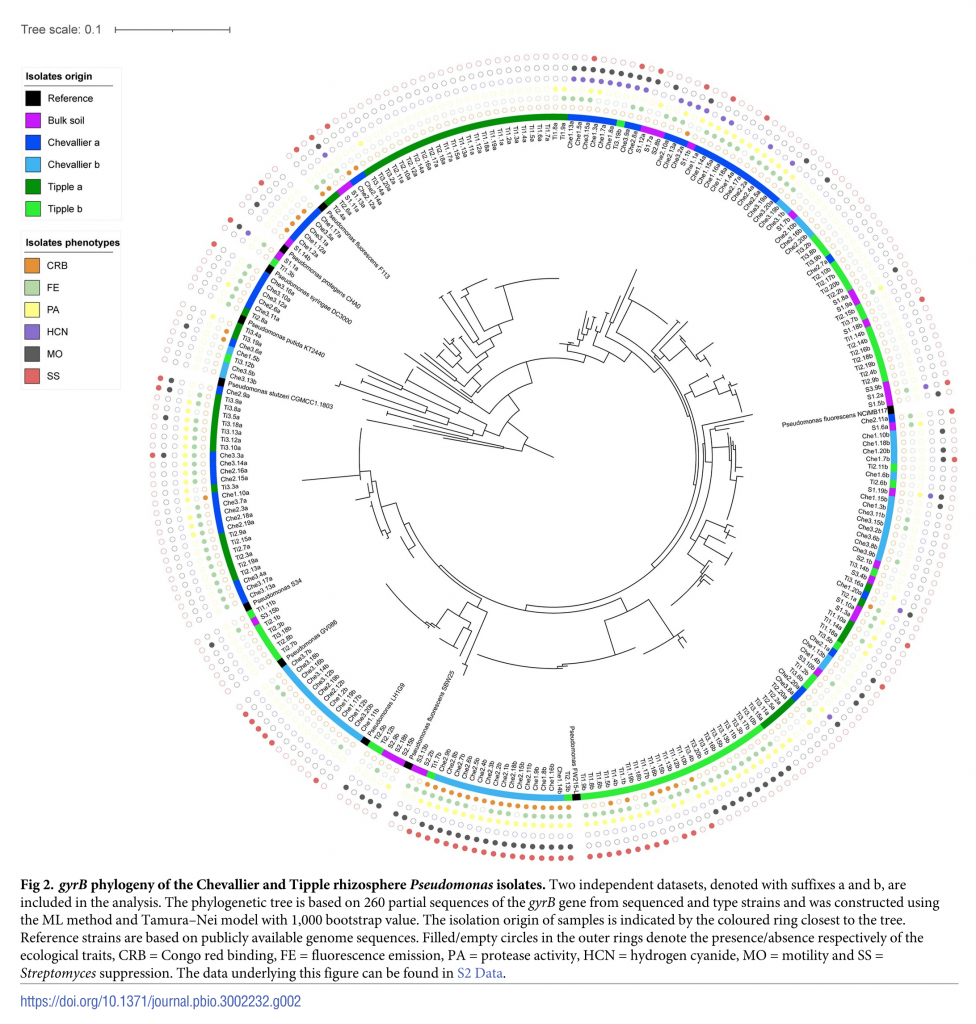
La abundancia de Pseudomonas fue diferente en ambas variedades, siendo más abundante en Tipple que en Chevalier y se realizaron aislados de este género a partir de la rizósfera de ambos genotipos y también del sustrato usado para su cultivo. Un total de 218 aislados fueron obtenidos y de cada uno se secuenció parte del gen gyrB para determinar la genotipificación. El árbol filogenético elaborado a partir de estas secuencias muestra la agrupación de los aislados por genotipo de cebada y tipo de sustrato utilizado (a o b). Los aislados pertenecientes a un mismo grupo tienen fenotipos similares, lo cual sugiere la selección por parte de la planta.
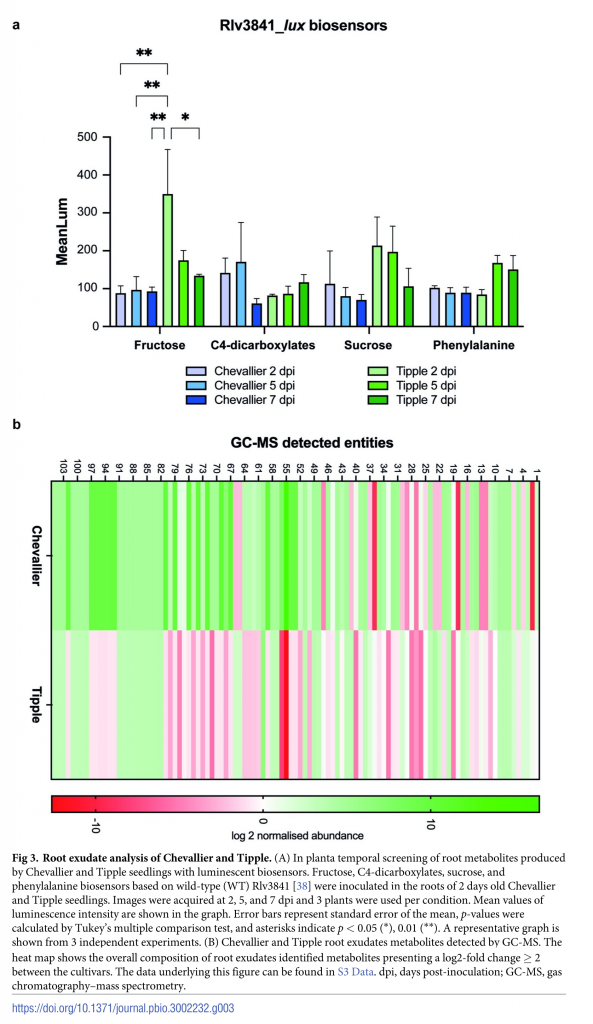
Para explorar los cambios en la composición del microbioma debido al genotipo de la planta se estudió la composición de exudados radiculares de Tipple y Chevallier utilizando biosensores luminiscentes para la detección de fructosa, sucrosa, dicarboxilatos de cuatro carbonos y fenilanania como miembros representativos de azúcares, ácidos orgánicos y aminoácidos. Las medidas se realizaron 2, 5 y 7 días después de la inoclación con el biosensor. La mayor diferencia se presentó en la detección de fructosa en Tipple para el primer tiempo medido. Mediante cromatografía de gases y espectrometría de masas se identificaron 105 compuestos con abundancia diferencial entre ambos genotipos, de los cuales únicamente 60 pudieron ser identificados. Chevallier presentó una mayor diversidad metabólica, mientras que Tipple mostró una mayor abundancia de D-glucosa, sugiriendo su papel en la estructuración del microbioma.
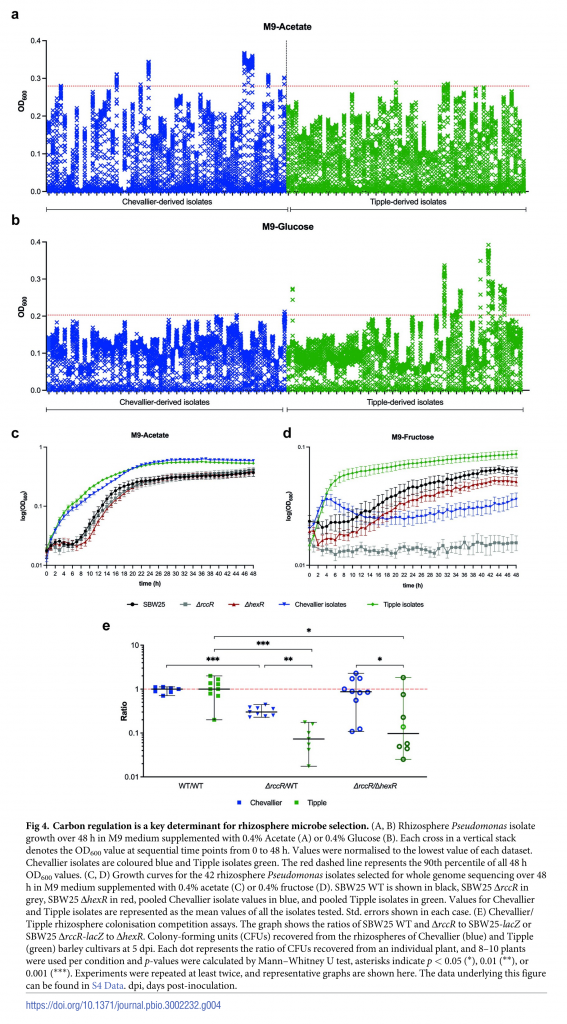
Para evaluar el papel de la composición de los exudados radiculares en la selección de aislados de Pseudomonas, se evaluó el crecimiento de los aislados de ambos genotipos en medio mínimo suplementado únicamente con una de dos fuentes de carbono: glucosa o acetato. 9 asilados de Chevalier presentaron densidad óptica por encima del percentil 90, respecto a 3 de Tipple. Por otra parte, al ser cultivados con glucosa, 10 aislados de Tipple se encontraron por encima del percentil 90, respecto a 3 de Chevalier. Estos datos fueron confirmados mediante la evaluación del crecimiento de asilados de cada genotipo, Pseudomonas fluorecens SBW25 y dos mutantes de la misma: ΔrccR y ΔhexR, las cuales muestran defectos en el crecimiento en glucosa y acetato respectivamente. El crecimiento en acetato presentó un patrón similar para todas cepas, aunque fue mayor para los aislados de Tipple y Chevallier. En cambio, el crecimiento en fructuosa fue mayor para los aislados de Tipple respecto a los de Chevallier, esta diferencia fue más drástica para la mutante ΔrccR. También se evaluó la capacidad de colonización de ΔrccR respecto a SBW25 en ambos genotipos, en los cuales mostró menor capacidad de colonización, pero fue mayor en Tipple. Un patrón similar se observó al comparar ambas mutantes, lo cual confirma la importancia de la presencia de hexosas para favorecer el crecimiento de ciertos aislados de Pseudomonas.
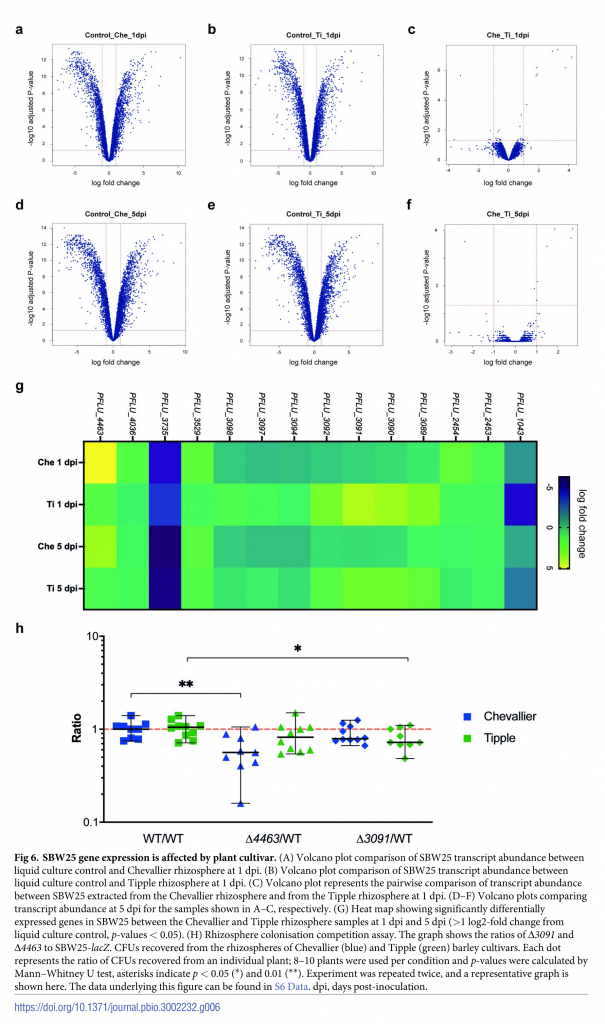
Se comparó la expresión genes de Pseudomonas fluorecens SBW25 en las raíces de Tipple y Chevallier además de un control en cultivo líquido, a 1 y 5 días después de la inoculación. La expresión de genes fue diferente entre el control y ambas rizósferas, pero bastante similar entre las rizósferas de los dos genotipos, con únicamente 14 genes diferencialmente expresados 1 día día después de la inoculación y 7 a los 5 días. Se seleccionaron dos genes de acuerdo con la abundancia de su expresión según el genotipo: PFLU_3091(más abundante en Tipple) y PFLU_4463 (más abundante en Chevallier), los cuales fueron deletados para evaluar su relevancia en la colonización de ambas rizósferas. ΔPFLU_4463 presentó una menor capacidad para colonizar la rizósfera en Chevallier, pero no en Tipple, mientras que ΔPFLU_3901 no presentó una disminución tan drástica en su capacidad para la colonización de Tipple.
Este trabajo resalta el papel de los exudados radiculares en la selección de microorganismos, favoreciendo el crecimiento de aquellos que cuentan con la capacidad metabólica para degradarlos, afectando su abundancia y la expresión de genes específicos.
Pacheco-Moreno, A., Bollmann-Giolai, A., Chandra, G., Brett, P., Davies, J., Thornton, O., Poole, P., Ramachandran, V., Brown, J. K. M., Nicholson, P., Ridout, C., DeVos, S., y Malone, J. G. (2024). The genotype of barley cultivars influences multiple aspects of their associated microbiota via differential root exudate secretion. PLOS Biology, 22(4), e3002232. https://doi.org/10.1371/journal.pbio.3002232



