Delimitando especies bacterianas con base en la taxonomía genómica
La base de datos de taxonomía genómica (GTDB, por sus siglas en inglés) es un esfuerzo de clasificación taxonómica para más de 150,000 organismos de los dominios Bacteria y Archaea (incluyendo genomas de oganismos no cultivables ensamblados a partir de secuencias metagenómicas). Este sistema se basa en las relaciones filogenéticas de las secuencias genómicas de estos organismos orientándose con base en clados monofiléticos tomando en cuenta las tasas de evolución relativas para cada linaje. Este trabajo reporta los avances de la GTDB en cuanto a la definición cuantitativa de especies tomando como base la identidad nucleotídica promedio (ANI, por sus siglas en inglés) y la fracción alineable entre los genomas de organismos estrechamente emparentados.
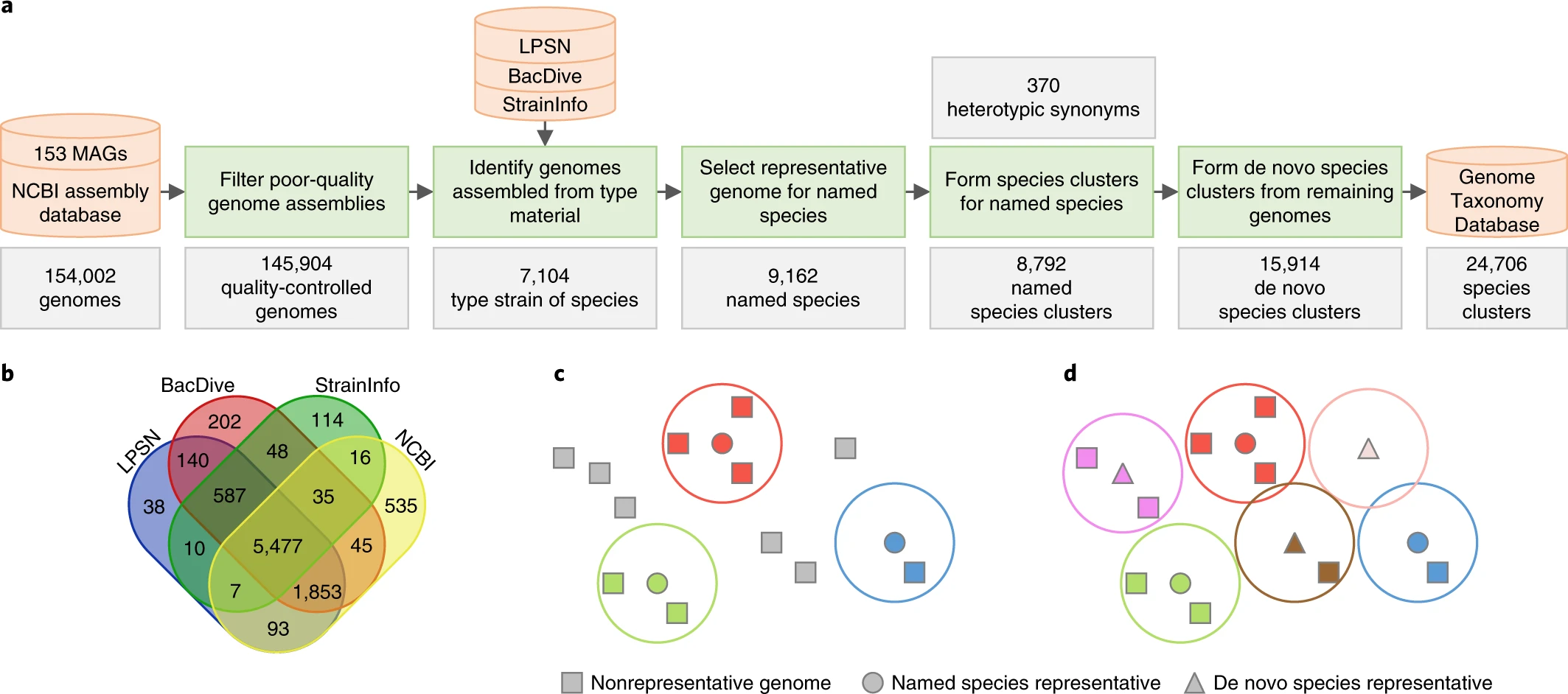
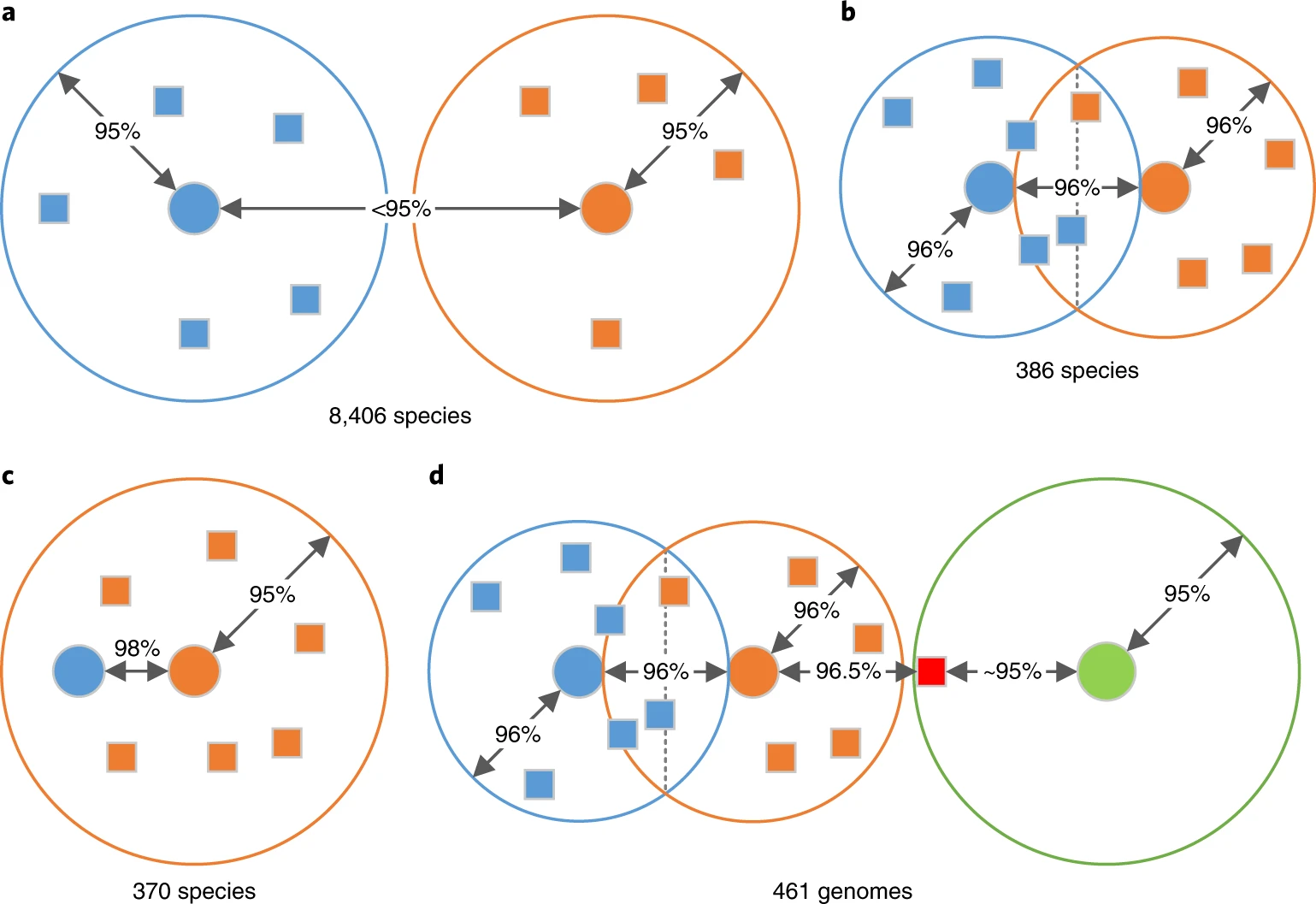
Parks y compañía crearon redes de genomas que se conectaban siempre que tuvieran un ANI de alrededor de 95% para formar racimos de especies. Posteriormente, para establecer los nombres para cada agrupamiento, revisaron si los racimos contaban con algún organismo tipo o de referencia para asignar el nombre y en su defecto se asignaron nombres provisionales. De los 24,706 racimos propuestos, 8,792 se basaron en nombres ya publicados y se tuvieron que dar 15,914 nombres provisionales para las nuevas especies de organismos no cultivables.
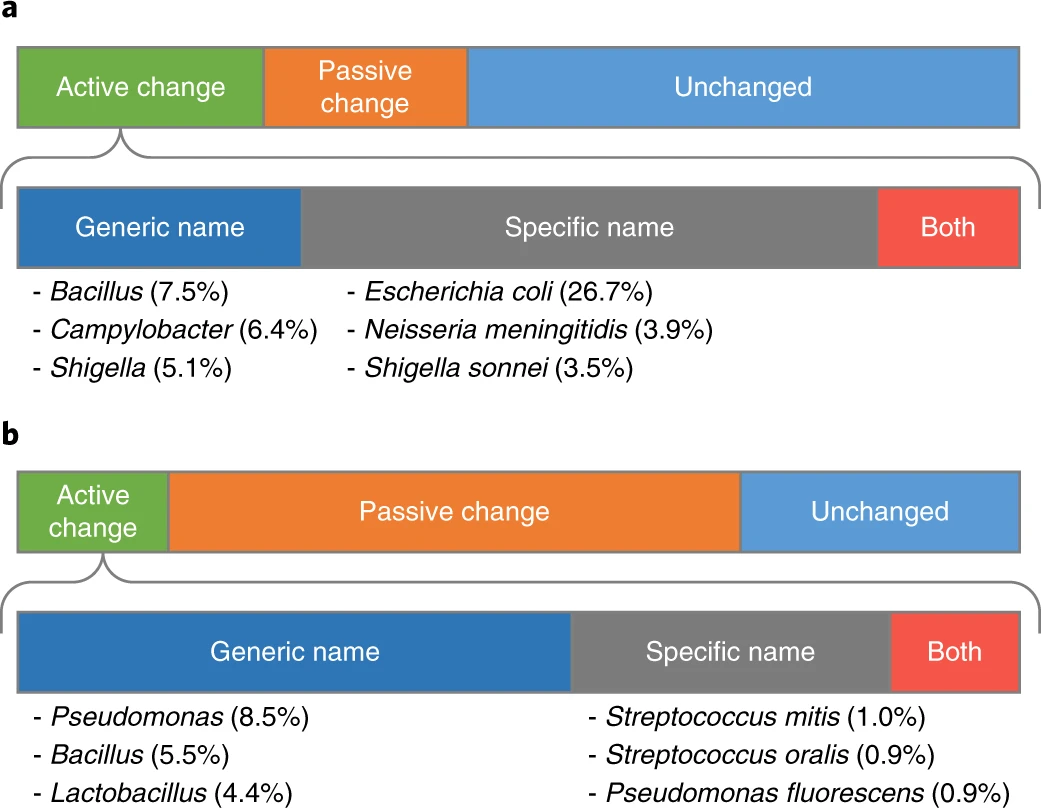
De los mas de 142,000 genomas con clasificación taxonómica en el NCBI, el 76% tuvo una especie asignada, de los cuales el 30% tuvo una asignación diferente en la GTDB. Esto se debe a reclasificaciones en los géneros Escherichia, Bacillus, Campylobacter y Shigella. Si observamos solo los organismos representativos, menos de la mitad de los racimos de especies con clasificación en el NCBI, fueron clasificados a nivel de especie. De estos, el 35% tuvo una clasificación diferente gracias a la resolución de géneros polifiléticos o a la normalización de géneros por tasas de cambio evolutivo diferentes. Estos cambios incluyen géneros ya reconocidos como polifiléticos como Pseudomonas, Bacillus y Clostridium.
Esta base de datos representa una herramienta sumamente útil para analizar genomas en donde la jerarquía taxonómica tiene un significado filogenético preciso. Además, facilita el trabajo con organismos no cultivables, de los cuales ha crecido nuestro conocimiento en los últimos años.
Parks, D. H., Chuvochina, M., Chaumeil, P. A., Rinke, C., Mussig, A. J., & Hugenholtz, P. (2020). A complete domain-to-species taxonomy for Bacteria and Archaea. Nature Biotechnology, 1-8.



