Distribución de las rutas de síntesis de ácido indol-3-acético dependiente de triptófano en bacterias usando análisis genómicos.
El ácido indol-3-acético (AIA) es una fitohormona crucial en el desarrollo de las plantas ya que, controla muchos procesos que incluyen la división celular, las respuestas a luz y gravedad. Muchas bacterias pueden sintetizar AIA y así promover el crecimiento vegetal, sin embargo, en bacterias el AIA sirve como una molécula de señalización. Este AIA es sintetizado mediante 4 rutas dependientes de L-triptófano. La vía indol-3-piruvato (IPA), triptamina (TPM), indol-3-acetonitrilo (IAN) e indol-3-acetamida (AIM). Esta última ruta es la mejor caracterizada en bacterias simbióticas. Algunas enzimas que participan en estas rutas han sido estudiadas y caracterizadas in vitro. Además, se sabe que muchas bacterias pueden tener más de una ruta de biosíntesis de AIA en su genoma como se ha observado en algunas Proteobacterias y Actinobacterias. Sin embargo, existe una escasez de información sobre el potencial biosintético del AIA en comunidades bacterianas. Por lo que en este estudio Zhang y colaboradores proponen el uso de un análisis genómico masivo para proporcionar información sobre la distribución filogenética de estas rutas. Para llevar a cabo dicho estudio, usaron más de 7000 genomas disponibles en las bases de datos y datos metagenómicos de los cuales se obtuvieron bins. El objetivo fue trazar todas las rutas de AIA en bacterias.
Para determinar la distribución de la ruta de biosíntesis de AIA en bacterias, se recuperaron 7282 genomas completos y en borrador; 114 de Acidobacterias, 923 de Actinobacterias, 281 de Bacteroidetes, 1705 de Firmicutes y 4259 de Proteobacterias. En todos estos genomas se buscaron las 4 rutas de biosíntesis de AIA. Encontraron que: Acidobacterias, Bacteroidetes y Firmicutes pueden sintetizar AIA mediante las rutas TPM e IAM. En proteobacterias ocurre vía IPA y TPM. El 50% de los genomas de Acidobacterias y el 30.8% de Actinobacteria son capaces de sintetizar AIA a partir de L-trp. En resumen, encontraron que las rutas de biosíntesis de IAM y TPM son las rutas más abundantes en los genomas bacterianos. Sin embargo, en la ruta IPA, se necesita de una 1-amino oxidasa, y en sus análisis no encontraron ortólogos de aminotransferasas específicas, esto es un hueco metabólico, por esta razón, examinaron la posibilidad de que las bacterias analizadas pudieran producir el AIA a través de intermediarios, es decir en donde sólo se necesita de un paso enzimático. Sus resultados muestran que la mayoría de los genomas analizados pueden sintetizar AIA a partir de intermediarios debido a que tienen algunas enzimas.
Recientemente, se había observado que más de una ruta de biosíntesis de AIA podría estar presente en un solo organismo, no obstante, no se había cuantificado cuántas y cuáles rutas eran las más frecuentes en los diferentes fila. Por lo que, en este estudio, se cuantificó el número de rutas presentes en los genomas analizados. Se encontró que; en 119 genomas, la mayoría del fila Proteobacteria, existen hasta tres enzimas que participan en la utilización de Trp. Mientras que en cinco genomas de Proteobacteria existen tres rutas completas: IPA, IAM y TPM, en los genomas de Firmicutes sólo se encontró una ruta de biosíntesis, además, se encontró que en este filo se puede producir AIA a partir de intermediarios.
La producción de AIA es una característica de bacterias promotoras de crecimiento vegetal, pero, se sabe muy poco sobre cómo las comunidades microbianas producen el AIA en entornos asociados con plantas como las rizósferas. Por esta razón, se cuantificó la abundancia de diferentes genes relacionados con la biosíntesis de AIA utilizando 27 metagenomas de rizósferas (RS) y 19 de rizoplano (RP) provenientes de la planta Setaria itálica (cola de zorra), una planta China emblemática. Encontraron que los genes más abundantes son los de las rutas de IAM y TPM, además su abundancia es diferente en RS y RP (Figura 1). Muchas de las enzimas que encontraron no pudieron ser asignadas a un grupo bacteriano conocido, por lo que es probable que existen otros organismos en los metagenomas que pueden sintetizar AIA y que no se han estudiado. Así mismo, encontraron muchas enzimas asociadas a Proteobacterias y Actinobacterias, por lo que estos fila participan en la producción de AIA en raíz.
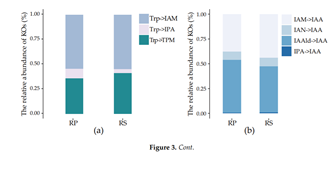
Figura 1. Quantification of the IAA synthesis related genes in root environments. The bar plots show the relative abundance of (a) Trp-catalyzing enzymes and (b) enzymes in the last step of IAA synthesis in rhizosphere (RS) and rhizoplane (RP), respectively (Modificado de Zhang et al. 2019).
Para comparar la capacidad de síntesis de AIA entre rizobacterias y los genomas que se recuperaron, se obtuvieron bins, 117 de Proteobacteria, 60 de Acidobacteria, 49 de Bacteroidetes, 29 de Firmicutes, y 24 de Actinobacteria. Se encontró que el 60% podrían sintetizar AIA a partir de L-Trp y el 92% a lo hacía a través de intermediarios. Además, los bins también presentaron diferentes rutas de síntesis de AIA. Por lo que con ello se demostró que las rizobacterias tienen un gran potencial para sintetizar AIA. También observaron que en las rizósferas las enzimas necesarias para la biosíntesis de AIA estaban casi ausentes en Firmicutes. Finalmente, dado que se ha observado que los genes involucrados en la biosíntesis de AIA se localizan en el cromosoma, utilizan un software para detectar cuales de estos genes podrían estar en plásmidos (Figura 2). Así encontraron que la mayoría de los genes que se encuentran en plásmidos provienen de Proteobacterias.
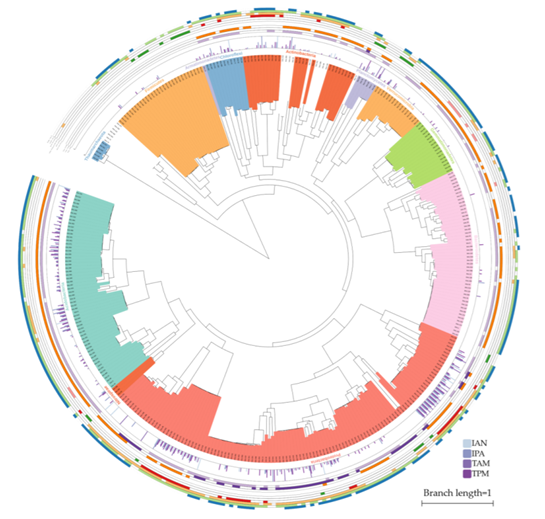
Figura 2. The phylogeny and distribution of IAA synthesis pathways in rhizobacteria. The tips of the tree are masked with different colors to indicate corresponding phylum. The histograms represent the percent of genes in each pathway located on plasmids. The three inner rings and six outside rings show the distribution of different Trp-catalyzing enzymes and capacities for synthesizing IAA from intermediates by different pathways, respectively. The blank space along the rings represent that no related genes or pathways are found in the rhizobacterium.
En conclusión, este es un ejemplo claro del uso de la metagenómica para analizar comunidades de rizósferas que sintetizan AIA, lo que podría contribuir a la colonización exitosa de las rizobacterias y al conocimiento de la distribución taxonómica de estas rutas para búsquedas de organismos selectos con potencial de promoción del crecimiento vegetal.
Referencia
Zhang, P., Jin, T., Kumar Sahu, S., Xu, J., Shi, Q., Liu, H., & Wang, Y. (2019). The Distribution of Tryptophan-Dependent Indole-3-Acetic Acid Synthesis Pathways in Bacteria Unraveled by Large-Scale Genomic Analysis. Molecules (Basel, Switzerland), 24(7), 1411.


