La sequía altera los perfiles transcripcionales y metabólicos del suelo
Con la finalidad de conocer los posibles efectos del cambio climático en los suelos, los autores de éste trabajo utilizaron suelos de pradera expuestos a desecación, sobre saturación de agua y condiciones ambientales de humedad (grupo control); para evaluar los efectos de la sequía y el exceso de húmedad en las comunidades de microorganismos del suelo, los genes que expresan y los metabolitos que producen. Además de los grupos de comparación anteriormente mencionados, se utilizaron muestras de tres distintos suelos, en las cuales se evaluó la diversidad taxonómica por medio de la secuenciación masiva del gen 16S rRNA, metatranscriptómica y análisis de metabolitos por medio de cromatografia de gases-espectrometria de masas.
En cuanto a la diversidad taxonómica, los análisis de ordenamiento mostraron distinguir diferencias entre los suelos sometidos a la desecación y el resto de las muestras, aunque cabe señalar que los tratamientos sometidos a saturación de agua no mostraron diferencias respecto a los controles. Al comparar los cotroles contra los tratamientos, el de sequía mostró un enriquecimiento en OTUs de los Phyla WS3, Chloroflexi y Actinobacteria; mientras que el grupo húmedo mostró enriquecimiento en algunos OTUs de Proteobacteria.

De manera similar a los resultados obtenidos al analizar los perfiles taxonómicos, los perfiles transcripcionales mostraron diferencias entre el grupo húmedo y el seco. Al asignar taxonómicamente los transcritos, se descurbió que la mayor parte correspondía al grupo de las Terrabacterias, que incluye Actinobacterias, Firmicutes, Cyanobacteria, Chloroflexi y Deinococcus-Thermus, aunque es importante señalar que la cantidad de transcritos asignados a éste grupo fue mayor en el tratamiento seco que en el húmedo. Profundizando en las diferencias debidas al tratamiento, se encontró en las muestras áridas una mayor cantidad de transcritos relacionados con el metabolismo de nucleótidos –

En análisis de los perfiles metabólicos permitio detectar 165 metabolitos, de los cuales solo se lograron identificar 70 en las bases de datos. Los análisis de diversidad beta permitieron distinguir algunas diferencias entre los tratamientos, sin embargo éstas diferencias sólo se volvieron notables cuando se consideró unicamente a los metabolitos con cambios entre los tratamientos, mostrando diferencias en los tratamientos expuestos a sequía. La desecación de los suelos provocó cambios en la abundancia de 15 metabolitos que incluyen azúcares y ácidos carboxílicos.
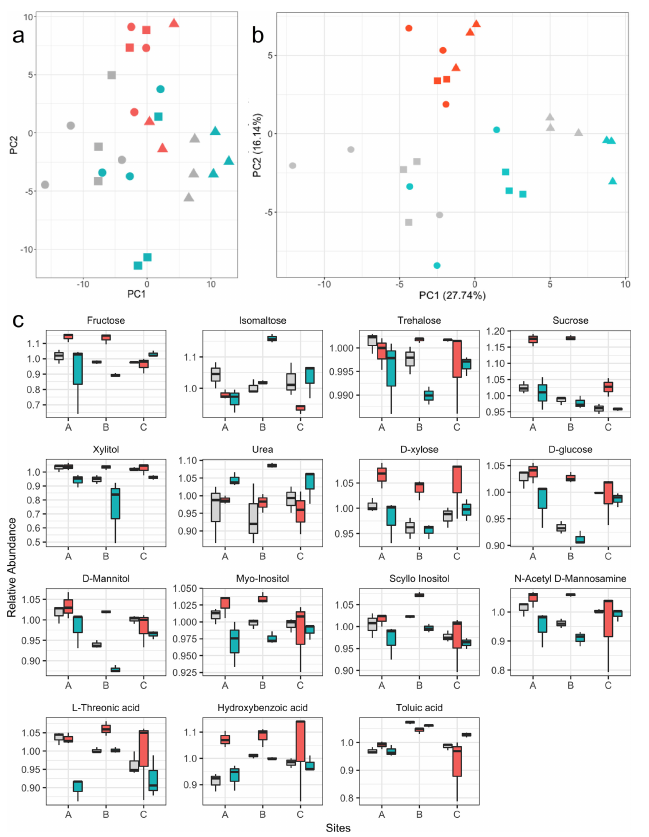
Finalmente, los autores del trabajo proponen la creación de redes de reacciones en las que se muestran las reacciones químicas y los metabolitos en función del tratamiento, resaltando los que se sobreexpresaban en condiciones secas o en condiciones húmedas. Entre los módulos relacionados con las condiciones secas, destacan algunas reacciones como la síntesis del osmoprotector trehalosa, la cual es síntetizada bajo dichas condiciones, pero degradada en los tratamientos húmedos. Con éste trabajo es posible conocer mejor las respuestas fisiológicas de los microorganismos del suelo a la sequía, para comprender un poco mejor los efectos que el cambio climático podría ocasionar en los mismos.

Referencia
Chowdhury, T. R., Lee, J. Y., Bottos, E. M., Brislawn, C. J., White, R. A., Bramer, L. M., … & Rice, C. W. (2019). Metaphenomic responses of a native prairie soil microbiome to moisture perturbations. MSystems, 4(4), e00061-19.


