Microbioma y resistoma de distintas superficies en un hospital.
En el contexto de la situación de los últimos años respecto a la problemática de resistencia a antibióticos, se tiene como objetivo estudiar los lugares donde la adquisición y dispersión de los genes involucrados en dichos mecanismos de resistencia pudieran representar mayor riesgo, y uno de los principales sitios son los hospitales, principalmente por la alta incidencia de enfermedades nosocomiales y por la adquisición de multi-resistencias de patógenos oportunistas, lo que intensifica la problemática. Alrededor de este contexto, Chng y colaboradores, publicaron en el presente mes de junio, los resultados de su investigación de metagenómica de muestras de 7 diferentes superficies de un hospital en Singapur.

Respecto a la taxonomía, los resultados los agruparon en dos comunidades: la comunidad tipo A (CTA), que es más diversa y que en general correponde a las superficies con mayor frecuencia de contacto, y la comunidad B (CTB), que básicamente corresponde al lavamanos y a los aspersores de la llave. En la CTA se observaron abundancias mayores de géneros asociados a piel de humano tales como Cutibacterium, Acinetobacter, Corynebacterium y Staphylococcus, y en la CTB Achromobacter, Bordetella y Methylobacterium.

En cuanto a los patrones de resistomas, también observaron una diferencia en la composición de genes de resistencia dependiendo del sitio, por ejemplo: oxa-23 y mecA, son genes que se encontraron en mayor abundancia en la CTA,y en CTB, se encontó mayor abundancia en los genes imp-1 y cme-1.
Además, utilizaron un enfoque con técnicas dependientes de cutivo, donde en medios enriquecidos y adicionados con 5 diferentes antibióticos (ampicilina, cloranfenicol, kanamicina, estreptomicina y tetraciclina), cultivaron las mismas muestras de los mismos sitios que habían utilizado para obtener las lecturas de los metagenomas. De estas mismas cajas obtuvieron el DNA metagenómico y lo secuenciaron (GridION), y obtuvieron los genomas de 69 especies incluyendo, por ejemplo, Bacillus, Pseudomonas y Staphylococcus.
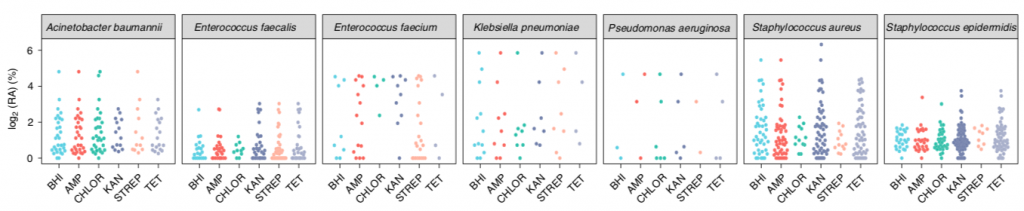
Posteriormente, con los mismos datos de metagenómica (no cultivables), hicieron la correlación de genes de resistencia a antibióticos para ver si se encontraban en plásmidos o en los cromosomas, para inferir cuáles se encontraban en mayor abundancia ya sea en transferencia vertical o en transferencia horizontal. Y algunos de sus resultados sí se han identificado en previos estudios como de importancia médica, por ejemplo, la co-ocurrencia de los genes fosB y mecA en cepas de Staphylococcus aureus, y también destacan que hallaron genes como fosA o mcr1, que confieren resistencia a antibióticos de último recurso. Mientras que en plásmidos encontraron co-ocurrencia entre genes que confieren resistencia a estreptomicina (strA-strB), a sulfonamida, a aminoglucósidos, a tretraciclinas y beta-lactámicos.

Concluyen que su trabajo resalta la importancia de que bacterias multi-resistentes permanezcan y se dispersen en ambientes hospitalarios y que a su vez los pacientes y personal médico se encuentren expuestas a ellas por medio de las superficies, y al igual mencionan que este tipo de estudios resaltan la importancia que se debe hacer para tomar aún más medidas de limpieza en los hospitales.
Chng, Kern Rei, et al. «Cartography of opportunistic pathogens and antibiotic resistance genes in a tertiary hospital environment.» Nature Medicine (2020): 1-11.

