En búsqueda de genes clave en Cellvibrio para la colonización de la rizósfera y el rizoplano
Cellvibrio es un género de Gammaproteobacterias que se ha encontrado en diversos ambientes como el suelo, el agua, el intestino de caracoles y raíces de plantas. En el caso de las raíces, se le ha encontrado enriquecido en muestras de rizósfera y de rizoplano del género Citrus, donde podría ser relevante puesto que posee la capacidad de degradar la pared celular vegetal. Los autores de éste trabajo emplearon un enfoque metagenómico y metatranscriptómico para develar los mecanismos que utiliza el género para la colonización de la rizósfera y el rizoplano. Para ello ensamblaroun un MAG (genoma reconstruido a partir de secuencias metagenómicas) de Cellvibrio y compararon la abundancia de los transcritos (detectados en el MAG) entre las muestras de rizósfera y rizoplano.
Para obtener el MAG se utilizó MetaBAT2, con lo que se obtuvo un genoma de 5.7 Mpb, compuesto por 41 contigs que fueron agrupados en 11 scaffolds. A grandes rasgos, la búsqueda por medio de BLAST mostró que existen varias regiones del genoma que no se encontraron en otras especies similares como C. mitxus y C. japonicus. Por otro lado, cabe señalar que el genoma fue más abundante en el rizoplano de Citrus que en su rizósfera.
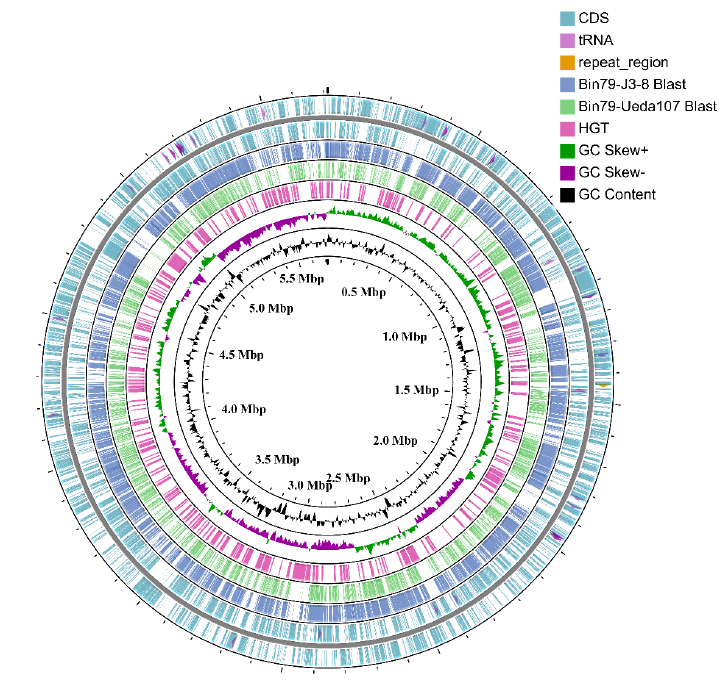
Figure 1 Circular representation of the Cellvibrio sp. Bin79 genome. The contents of the feature rings (starting with the outermost ring) are as follows: Ring 1 and Ring 2, features from the forward and reverse strands, respectively, CDSs are drawn as arcs, and tRNAs and repeat regions are drawn as arrows; Ring 3 (Cellvibrio mixtus subsp. mixtus J3-8) and Ring 4 (Cellvibrio japonicus Ueda107) show BLAST comparison results (BLASTN, E-value 1e−20) with the Bin79 genome, the BLAST comparison results are drawn with partial opacity, and darker regions indicate that multiple hits to the corresponding sequence region were observed; Ring 5 shows the genomic regions acquired via horizontal gene transfer; Ring 6 shows GC skew; and Ring 7 shows GC content. The plot was drawn using the CGview server.
Para comparar los transcritos asociados a rizósfera y rizoplano, se hizo un mapeo contra el genoma ensamblado de Cellvibrio. La expresión de genes para la síntesis de ácido indol acético no mostró diferencias entre rizósfera y rizoplano. No obstante, los análisis estadísticos muestran que la rizósfera sobreexpresa genes que codifican enzimas para la degradación de celulosa, arabinano y xilano. Mientras que en el rizoplano se expresan más genes para la captura y utilización de glucosa, así como para la degradación de quitina. Las quitinasas en el rizoplano podrían ser relevantes para la degradación de hongos patógenos, por lo que podrían ser benéficas para la salud de las plantas. De acuerdo con las categorías de COG, tanto «Síntesis y transporte de aminoácidos» como «Biogénesis y síntesis de pared y envolura celular» son más abundante en la rizósfera. Algunos de estos genes participan en la síntesis y exporte de lipopolisacáridos. Por su parte, el rizoplano subreexpresa genes de «Movilidad Celular», que incluyen algunos implicados en quimiotaxis y síntesis del flagelo. De acuerdo con estos resultados, los autores sugieren que en la rizósfera las bacterias tienden a formar biofilm, mientras que en el rizoplano las bacterias se mueven para capturar nutrientes liberados por las raíces de las plantas.
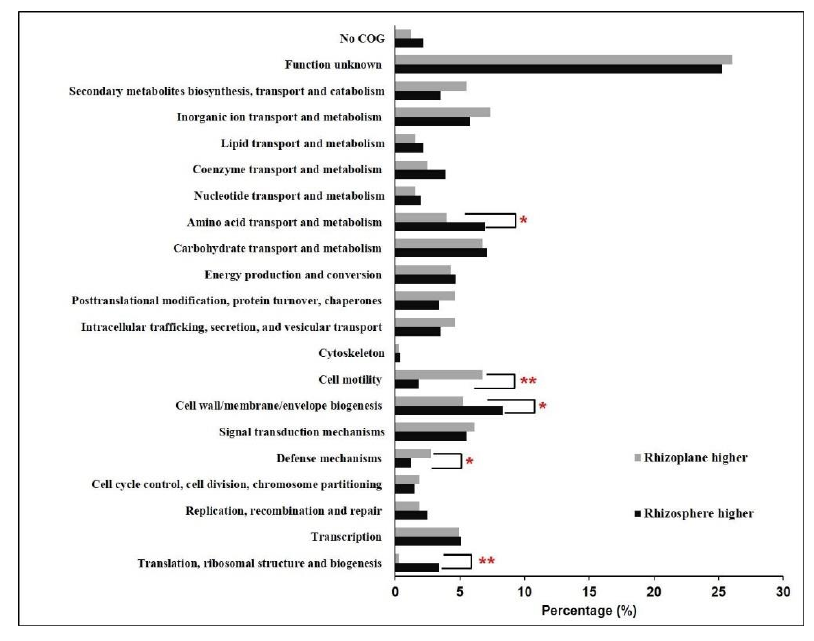
Figure 4 Functional distribution of the Cellvibrio sp. Bin79 differentially expressed genes between the rhizoplane and rhizosphere. *, p < 0.05; **, p < 0.01, determined by one-tailed Fisher’s exact test.
Por otro lado, el rizoplano muestra una mayor expresión de genes de resistencia a multidrogas y bombas de flujo, que las podrían hacer resistente a un amplio espectro de antibióticos. Se encontraron los genes ohr y su regulador ohrR, que permiten resistir a hidroperoxidos. Algo a destacar de dicho hallazgo es que ni ohr ni ohrR se han encontrado en otros Cellvibrios disponibles en las bases de datos, pero tienen homólogos en Reinhermaniera, por lo que pudieron ser adquiridos por transferencia horizontal.
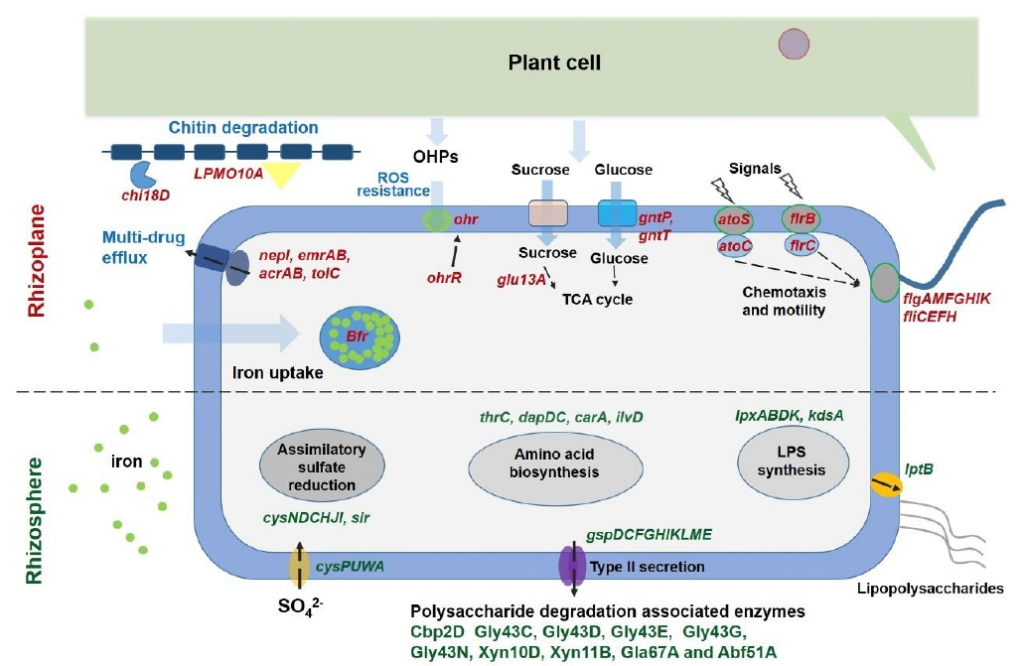
Figure 3 Overview of the genes with expression level differing between rhizoplane and rhizosphere habitats. Genes with higher expression level in the rhizoplane niche are red colored, while genes with higher expression level in the rhizosphere habitat are dark green colored. Iron is indicated by green circles, chitin is indicated by blue line-linked rectangles. The arrows denote influx and efflux of compounds.
Finalmente, se realizó un análisis de genómica comparativa del MAG respecto a otros seis genomas de Cellvibrio. El genoma núcleo del génerocomprendió 2018 grupos de ortólogos, mientras que el pangenoma 9210. Se identificaron varios eventos de ganancia de genes que podrían haber sido adquiridos mediante transferencia horizontal, como el caso del gen traG, implicado en la conjugación. Este tipo de estudios de genómica comparativa es relevante porque puede ayudar a identificar genes de especies emparentadas que les permiten vivir en diferentes ambientes, algunos de los cuales se pueden expresar diferencialmente, como sucede en la rizósfera y el rizoplano.
Referencia
Zhang, Y., Xu, J., Wang, E., & Wang, N. (2020). Mechanisms underlying the rhizosphere-to-rhizoplane enrichment of cellvibrio unveiled by genome-centric metagenomics and metatranscriptomics. Microorganisms, 8(4), 583.



