La influencia del suelo de origen en la estructura del microbioma de raíz es mayor que el tipo de cultivar de tomate
En este trabajo se preguntan la influencia del suelo de origen y diferentes cultivares de tomate en la estructuración del microbioma de rizosfera de tomate, esto lo realizan mediante el cultivo de 11 diferentes cultivares de tomate en un mismo suelo y un mismo cultivar de tomate en 7 suelos distintos, 2 de origen comercial con alto contenido nutrimental y 5 de origen natural. Para el experimento de cultivares tomaron 11 cultivares y los crecieron en suelo de césped (JX), en el otro experimento de suelos usaron un cultivar Meiguodahong 168 (MG) y lo cultivaron en 7 suelos.
Los phyla dominantes en todas las rizosferas fueron Proteobacteria, Bacteroidetes, y Acidobacteria, seguido por Verrucomicrobia, Planctomycetes, Actinobacteria y Gemmatimonadetes. Sin embargo entre muestras por cultivares, incuyendo entre tomate y var. cerasiforme no encontraron diferencias significativas en la compocición por grupos taxonómicos ni diversidad alfa, mientras que entre diferentes suelos sí fueron significativas.
Figure 1. The composition and relative abundance of the major bacterial taxa and the 100 most abundant operational taxonomic units (OTUs) in the tomato rhizosphere microbiota. (A,C) The composition and relative abundance of major bacterial orders in tomato cultivar samples (A) or soil samples (C). Each bar represents the average value of three replicates in each sample group. (B,D) Heatmap depicting the 100 most abundant OTUs in the microbiota of tomato cultivar samples (B) or soil samples (D). Dendrogram links and distances between OTUs do not depict phylogenetic relationships; they are based on the number of reads (log-transformed) of OTUs within the samples. The legend and scale in the upper right corner of the figure show the colors in the heatmap associated with the relative abundance of OTUs (cluster of variables on the Y-axis) within each plant and soil sample (X-axis clustering). Tomato cultivar experiment: Xinzhongshu No. 4 (XZ), Huangshoutao (HT), Tiancheng (TC), Meiguodahong 168 (MG), Huapiqiu (HP), Huangshengnvguo (HS), Huangzhenzhu (HZ), Qiaokeli (QK), Yingtao (YT), Ziwucai (ZW) and Ziyixiannv (ZY); soil experiment: commodity organic nutritional soil (CF), potted plant nutrient soil (HF), vegetable field soil (DM), agricultural field soil (HQ), campus lawn soil (JX), forest soil (QS) and garden soil (XC).
La diferencia de composición entre comunidades depende del suelo de origen
Para mostrar el grado en el que los genotipos de las plantas y los suelos impactaron las comunidades bacterianas de la rizosfera de las raíces del tomate, se combinaron los datos de los experimentos con los cultivares de tomate y los del suelo, incluidos los de todos los cultivares y muestras de suelo.
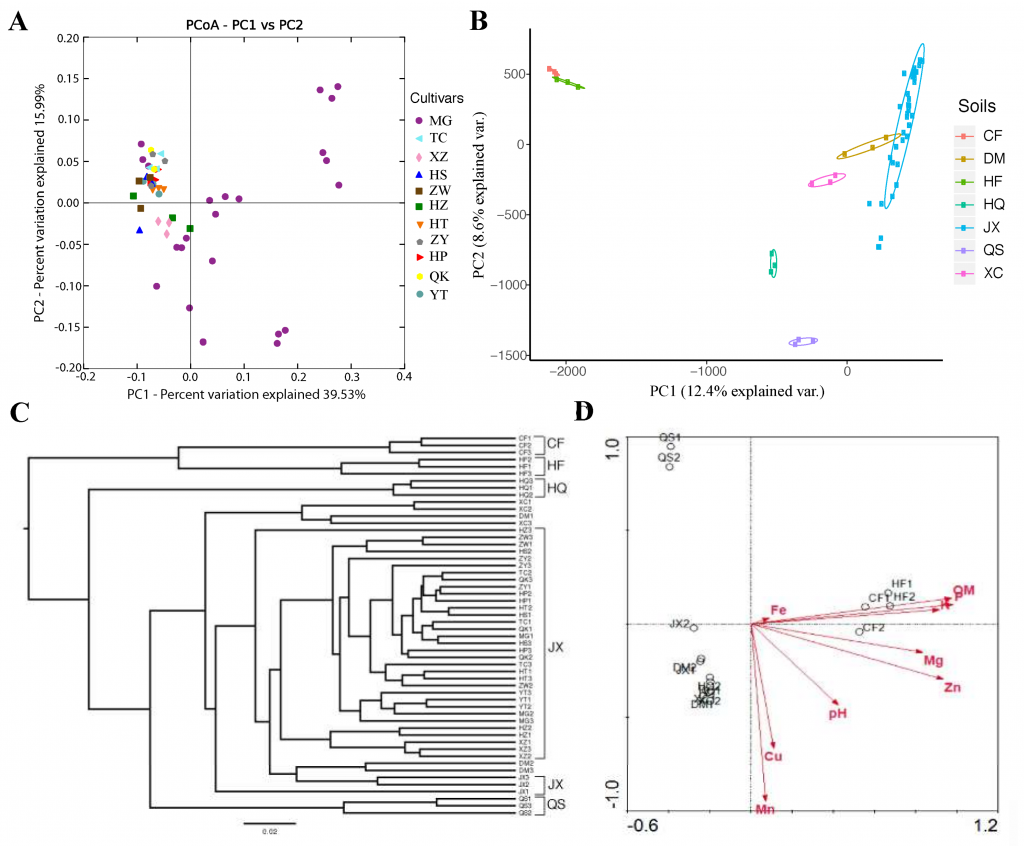
Figure 2. Beta diversity analysis to estimate the dissimilarity and similarity of bacterial community compositions among the cultivars and soil samples. (A) Principal coordinate analysis (PCoA) derived from the dissimilarity matrix of weighted UniFrac distances based on the combined data of the cultivars and soil samples. (B) Principal component analysis (PCA) showing sample grouping based on soil source from the combined data of the cultivars and soil samples. (C) Weighted UniFrac-based hierarchical cluster analysis of bacterial community composition based on the combined data of the cultivars and soil samples. (D) Canonical correspondence analysis (CCA) showing the main soil biochemical factors that affected the assembly and composition of the rhizobacterial communities based on the combined data of the cultivars and soil samples. Tomato cultivar experiment: Xinzhongshu No. 4 (XZ), Huangshoutao (HT), Tiancheng (TC), Meiguodahong 168 (MG), Huapiqiu (HP), Huangshengnvguo (HS), Huangzhenzhu (HZ), Qiaokeli (QK), Yingtao (YT), Ziwucai (ZW) and Ziyixiannv (ZY); soil experiment: commodity organic nutritional soil (CF), potted plant nutrient soil (HF), vegetable field soil (DM), agricultural field soil (HQ), campus lawn soil (JX), forest soil (QS) and garden soil (XC).
A partir de los datos de composición y abundancia de las comunidades microbianas calculan la disimilitud de Bary-Curtis y con ello la diversidad entre comunidades, gracias a ello se pueden distinguir los factores que más aportan a explicar la cercanía de las comunidades, siendo que los diferentes cultivares se parecen mucho entre sí, mientras que en un PCA que muestra solo la variación basada en componentes estadísticos de las abundancias de los OTUs muestran comunidades distintas entre las rizosferas del experimento de diferentes suelos (con las comunidades más divergentes que fueron cultivadas en suelos ricos en nutrientes: suelo nutritivo orgánico comercial (CF), suelo nutritivo para plantas en maceta (HF)).
Esto refuerza la conclusión: independientemente del método, el tipo de suelo domina la estructuración del microbioma.
Así mismo, cuando realizar un dendrograma de Cluster jerárquico basado en UniFrac ponderado por cercanía filogenética (usa la distancia de disimilitud entre comunidades y agrupa las muestras de forma sucesiva en clústeres de mayor similitud) que es un árbol de agrupamiento jerárquico que muestra qué tan similares son las muestras entre sí, y éstas son más cercanas por el suelo de origen que por el genotipo, lo cual se observa claramente en todas las muestras de los cultivares en suelo de césped (JX).
La posible explicación de las diferencias de las comunidades de los suelos es debido a los factores físico-químicos del suelo (la materia orgánica, el P y el K fueron los principales factores ambientales del suelo) son los que mejor explican la variación en las comunidades. Adicionalmente estas diferencias de las comunidades parecían muy marcadas por el tipo de suelo, ya que usaron dos suelos comerciales que son ricos en nutrientes contra suelos naturales, y tanto las comunidades por sí mismas como por la composición de nutrientes termina separando fuertemente estos dos grupos de suelos, marcando que estas características son determinantes en la estructuración del microbioma.
A pesar de estas diferencias, el análisis del microbioma núcleo muestran que hay OTUs que son constantemente reclutados y enriquecidos en la rizosfera del tomate, mostrando que aunque las comunidades puedan cambiar su estructura, estos OTUs son muy importantes para el microbioma del tomate, esto debido a la atracción que pueda ejercer el tomate para reclutar estos microorganismos con el fin de promover actividades microbianas que puedan mejorar su aptitud en ambientes de suelo variables.
Referencia:
Cheng, Z., Lei, S., Li, Y., Huang, W., Ma, R., Xiong, J., Zhang, T., Jin, L., Haq, H. ul, Xu, X., & Tian, B. (2020). Revealing the Variation and Stability of Bacterial Communities in Tomato Rhizosphere Microbiota. Microorganisms, 8(2), Article 2. https://doi.org/10.3390/microorganisms8020170



