Genes de Sphingobium involucrados en la degradación de saponinas
Las saponinas son un grupo de compuestos exudados por las plantas, las cuales pueden ser de tipo esteroide u oleanano dependiendo de si el precursor es colesterol o ß-amirina, respectivamente. Estas moléculas son tóxicas para algunos microorganismos, sin embargsaponinaso; en la rizósfera pueden ser degradadas por bacterias presentes en el suelo, lo cual podría estar relacionao con la colonización de la planta. En este trabajo, se identificaron enzimas presentes en el género Sphingobium involucradas en la degradación de saponinas de tipo esteroide.
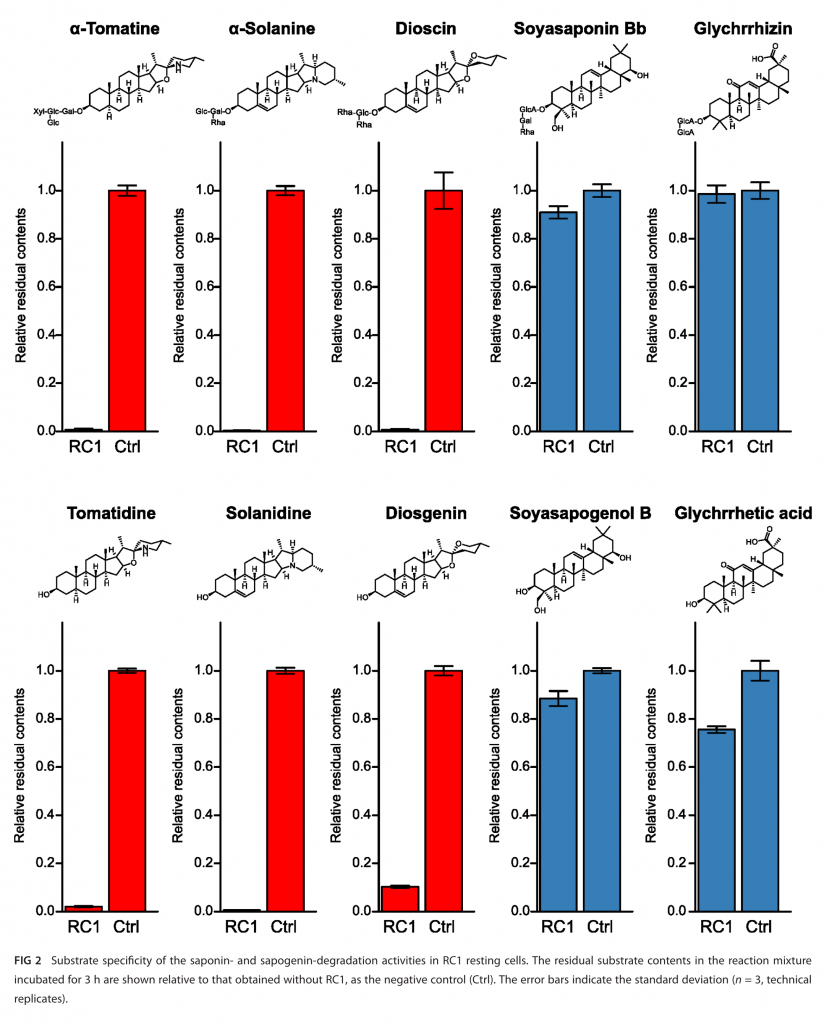
Se utilizó una cepa de Sphingobium aislada de raíces de tomate (RC1), la cual tuvo mayor capacidad de degradar saponinas de tipo esteroide (rojo), mientras que su actividad contra saponinas de tipo oleanano (azul).
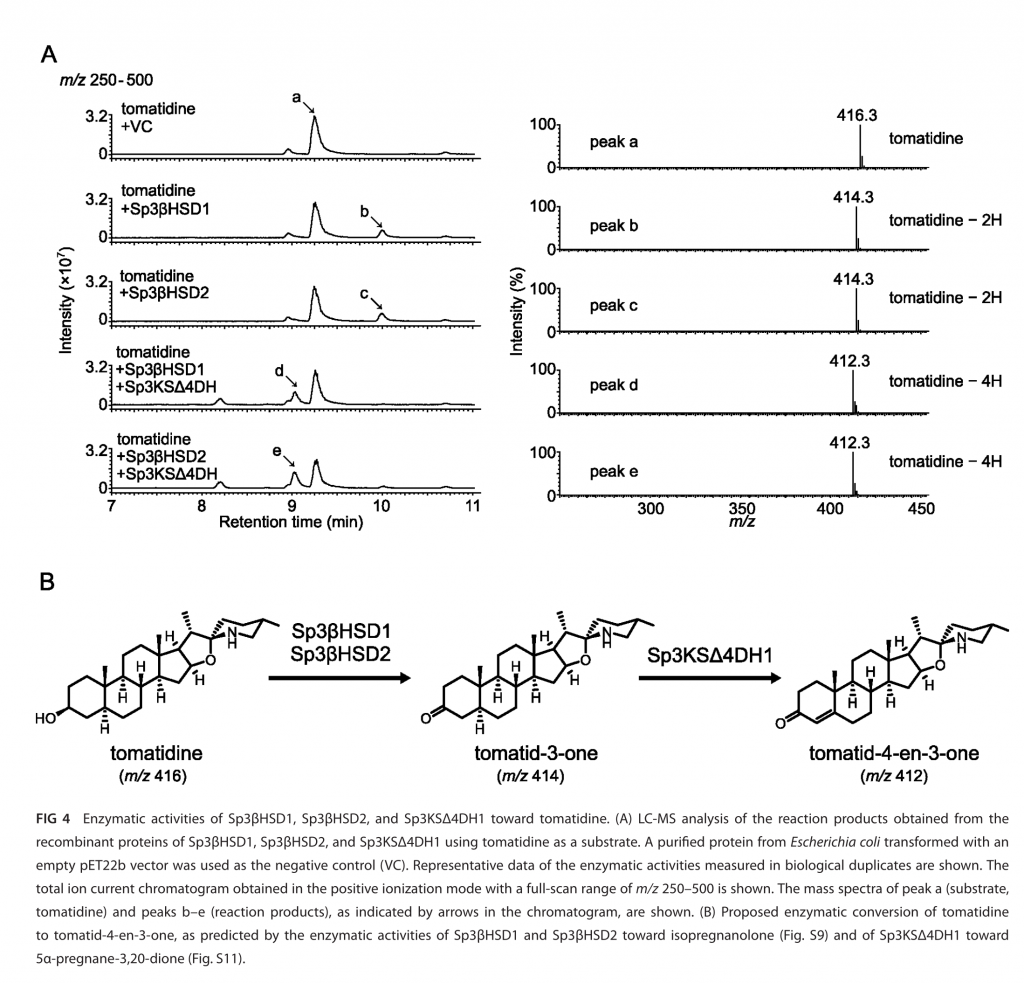
Utilizando el genoma de RC1, se buscaron deshidrogenasas que podrían participar en la oxidación del hidroxilo ubicado en el carbono 3 (C3) de las saponinas derivadas de colesterol. Se encontraron dos 3β-hydroxiesteroide deshidrogenasa (3βHSD), las cuales llamaron Sp3βHSD1 y Sp3βHSD2. El siguiente paso en la degradación de las saponinas consiste en la deshidrogenación del enlace entre los carbonos C5 y C6, lo cual es llevado a cabo por la 3-cetoesteroide-Δ4-deshidrogenasa (3KSΔ4DH). En el genoma de RC1 se encontró un candidato de esta enzima, denominado Sp3KSΔ4DH1. Posteriormente se evaluó la activdad enzimática de los candidatos en presencia de tomatidina y se analizaron los productos de reacción mediante cromatografía líquida acoplada a espectrometría de masas. Sp3βHSD1 y Sp3βHSD2 tuvieron la misma actividad sobre la tomatidina, generando picos (b y c) con un m/z consistente con tomatid-3-ona. Al incubar cualquiera de las dos hydroxiesteroide deshidrogenasas con Sp3KSΔ4DH1, se formó un nuevo pico (d y e), consistente con tomatid-4-en-3-ona. Estos resultados indican la participación de las enzimas de RC1 en los primeros pasos de la degradación de saponinas.
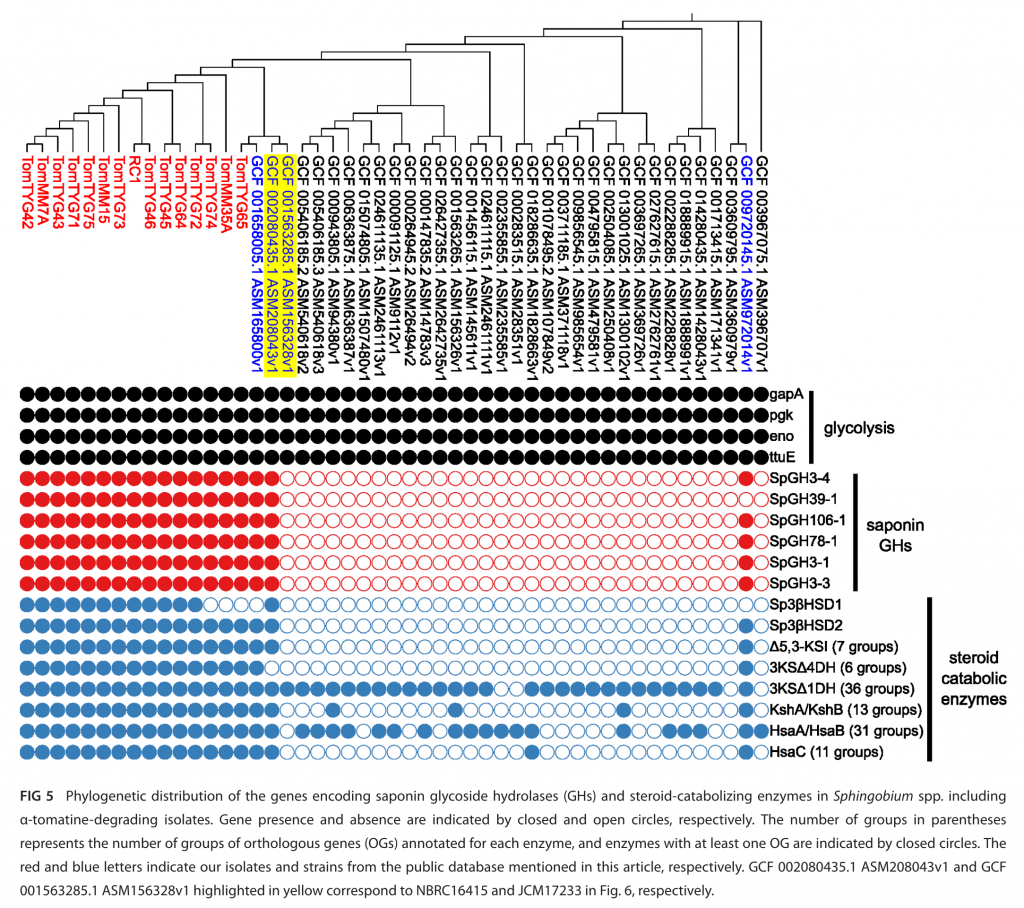
Utilizando cepas de Sphingobium aisladas de suelos tratados con tomatina y de raíces de tomate, así como genomas disponibles en bases de datos, se analizó la distribución filogenética de los genes identificados en RC1 que están involucrados en la degradación de las saponinas y de glucósido hidrolasas (GH) que convierten a las saponinas en sus respectivas sapogeninas para su posterior degradación. Aquellos genomas que no provienen de suelos tratados con tomatina o de raíces de tomate carecen de la mayoría de las enzimas involucradas en la degradación de saponinas
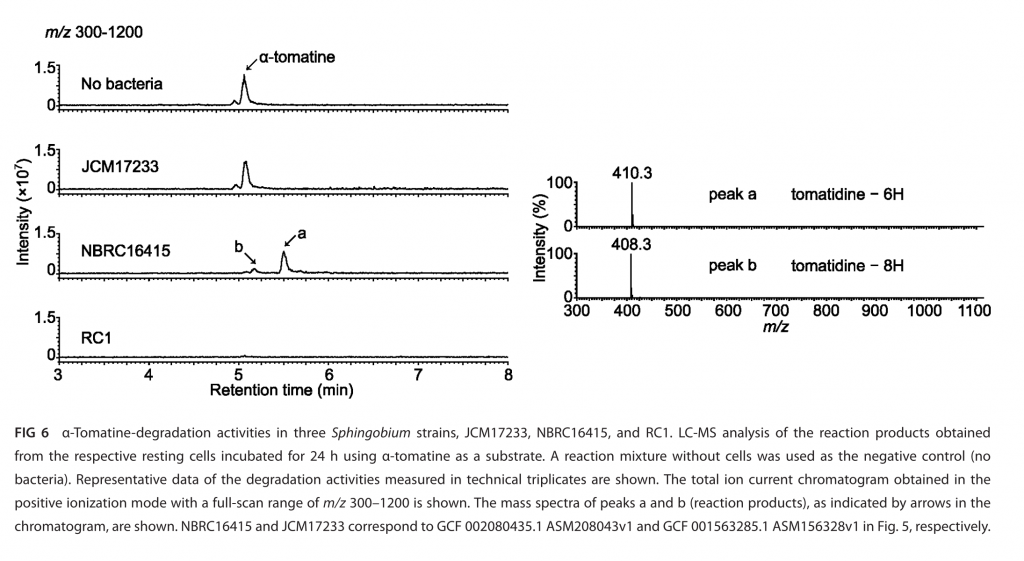
Para determinar si la presencia de los genes permite la degradación de saponinas, se incubaron células de RC1 y de NBRC16415 (GCF 002080435.1 ASM208043v1) y JCM17233 (GCF 001563285.1 ASM156328v1) en presencia de tomatina. NBRC16415 cuenta con un conjunto de genes similar a RC1 comparado con JCM17233 que carece de la mayor parte de los genes involucrados en la degradación de saponinas. De este modo, JCM17233 no tuvo actividad sobre la tomatina, mientras que NBRC16415 no pudo degradar completamente el mismo compuesto, probablemente debido a la ausencia de 3KSΔ4DH.
Estos resultados indican el inicio de la ruta a través de la cual miembros del género Sphingobium degradan saponinas. Es necesario describir los pasos subsecuentes para entender cómo las bacterias utiizan moléculas obtenidas por las plantas.
Referencia
Nakayasu, M., Takamatsu, K., Kanai, K., Masuda, S., Yamazaki, S., Aoki, Y., Shibata, A., Suda, W., Shirasu, K., Yazaki, K., & Sugiyama, A. (2023). Tomato root-associated Sphingobium harbors genes for catabolizing toxic steroidal glycoalkaloids. mBio, 14(5), e00599-23. https://doi.org/10.1128/mbio.00599-23


