¿El monocultivo tiene efectos en la susceptibilidad de infección en tomates y sus rizosferas?
Este estudio investiga cómo el cultivo sucesivo de tomate puede inducir una supresividad natural del suelo contra Ralstonia solanacearum, el agente causal de la marchitez bacteriana. Los autores plantean que los cambios inducidos en el microbioma del suelo y la rizosfera a lo largo de varios ciclos de cultivo generan comunidades microbianas capaces de restringir el desarrollo del patógeno.
Para probar esta hipótesis, realizaron múltiples ciclos de cultivo de tomate en condiciones controladas, comparando suelos previamente cultivados
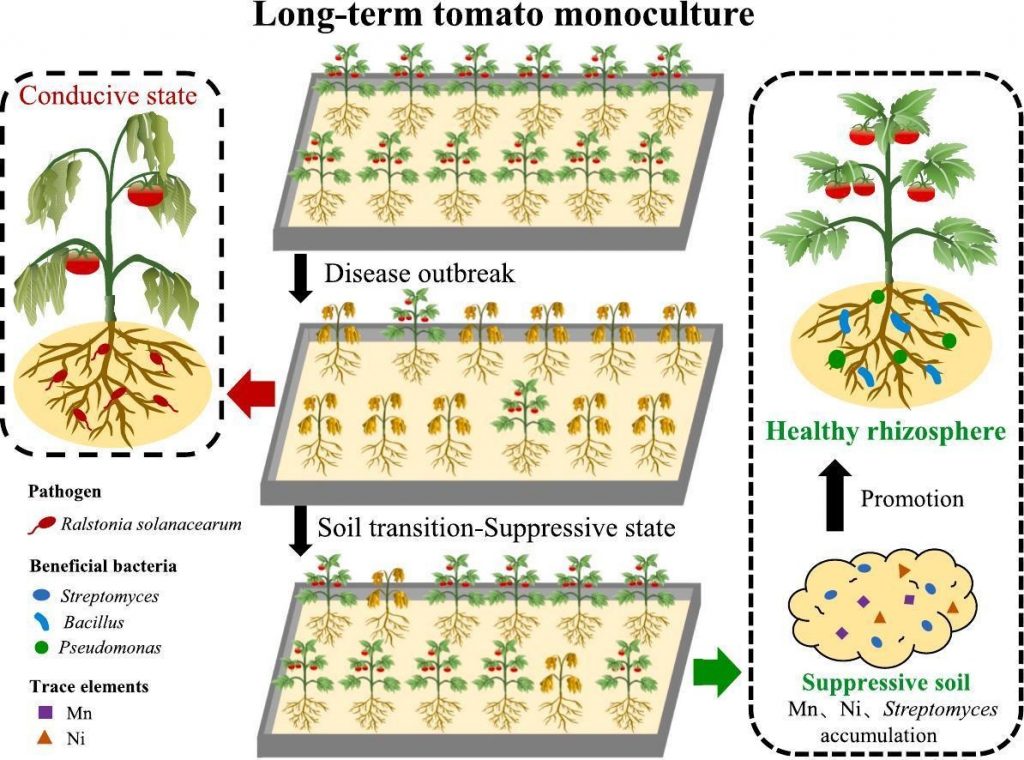
Los resultados indican que los suelos cultivados mostraron una disminución significativa de la enfermedad, acompañada de cambios estructurales en la comunidad bacteriana, incluyendo el enriquecimiento de ciertos taxones presumiblemente benéficos como Streptomyces.
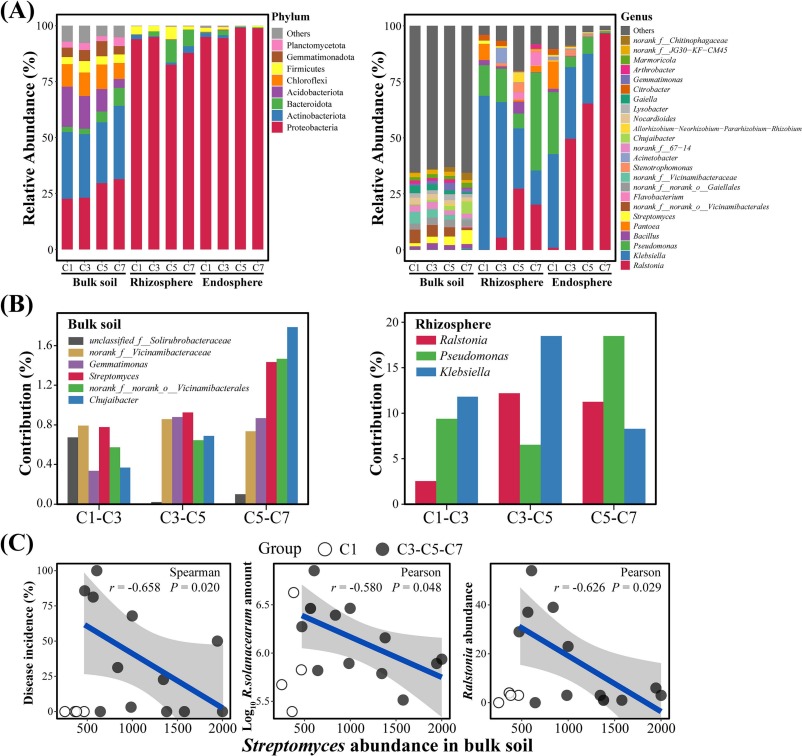
Fig. 2. Shifts in the bacterial community composition associated with changes in the disease incidence during tomato monoculture. Shown are: (A) the bacterial community composition at the phylum and genus levels, (B) taxa significantly contributing to the variation of bacterial communities across monocropping cycles based on the Similarity Percentage Analysis (SIMPER), and (C) the correlation between the abundance of Streptomyces and bacteria wilt incidence, as well as pathogen levels in bulk soil. “C1”, “C3”, “C5”, and “C7” represent the first, third, fifth and seventh monocropping cycles.
Aunque Streptomyces parece ser un biomarcador del suelo asociado con baja incidencia de Ralstonia, los datos no demuestran que controle la infección dentro de la planta (rizosfera o endosfera).
Por tanto, centrarse tanto en su abundancia podría ser un caso de sobreinterpretación funcional basada solo en correlación en un compartimento.
Aunque Streptomyces aparece como un predictor importante en el modelo de random forest, eso no significa que sea la causa principal de los efectos sobre Ralstonia o micronutrientes.
Podría ser simplemente un indicador de un entorno microbiano más complejo, o correlacionarse con otros factores como tipo de suelo, comunidad completa, o manejo agrícola o la propia disminución de la enfermedad.
La Figura 4A muestra qué tan importante es cada factor edáfico para predecir el enriquecimiento de Streptomyces y si su abundancia se asocia positiva o negativamente con ese valor.
Las barras que van hacia abajo indican que ese factor está negativamente correlacionado con el enriquecimiento, y el modelo lo detecta como útil. Desde mi punto de vista este análisis es mas útil si se analiza con el avance de la enfermedad del tomate, que es la principal variable de respuesta que los autores están observando.
A lo largo de estos ciclos de cultivo obtienen suelos que después etiquetan como S y C dependiendo del fenotipo de las plantas y el %de incidencia de enfermedad.
En invernadero se proponen cultivar tomates usando suelo etiquetado como «supresor de enfermedades» (S) y suelo «propicio para la enfermedad» (C) que usan en conjunto con la inoculación de la propia Rastonia (+R). De entrada el suelo supresor S parece producir mejor salud en las plantas que el suelo propicio C, y cuando son inoculadas con +R resultan con mejor fenotipo que las plantas del suelo propicio, aunque no con diferencias significativas cuando están inoculadas en el % de incidencia de la enfermedad y cantidad de Rasltonia. Además mencionan que la diversidad alfa es mayor en el suelo S, y la diversidad beta que aunque son suelos distintos en composición, al final se terminan pareciendo pero siguen siendo distintos.
Creo que sería relevante que incluyeran el contenido de la endosfera en estos análisis.
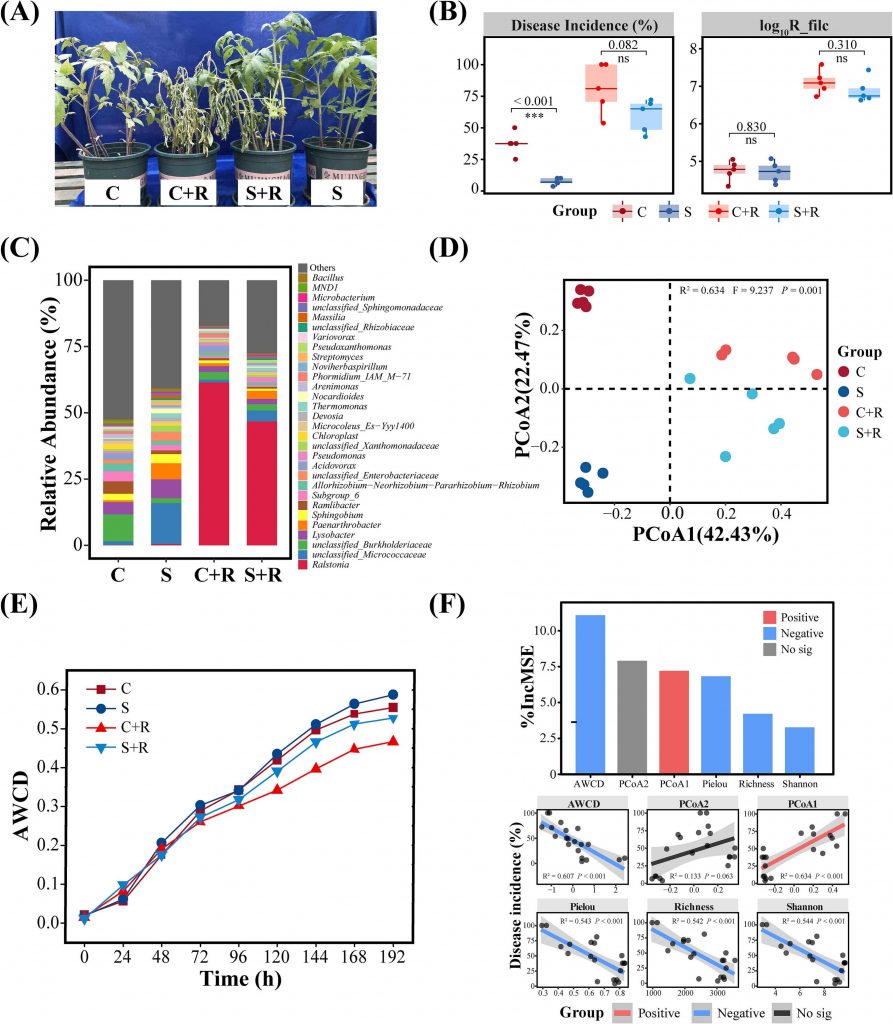
Fig. 5. The incidence of bacterial wilt, pathogen levels, and microbiome changes in the rhizosphere of tomato plants grown in disease-conducive (C) and disease-suppressive (S) soils without and with R. solanacearum (R) (greenhouse trial 1). (A) The appearance of 2-month-old tomato plants. (B) Disease incidence (left) and pathogen levels (right) in the tomato rhizosphere. (C) Composition of rhizosphere bacterial communities in different treatments. (D) Principal Coordinate Analysis (PCoA) of bacterial communities from the tomato rhizosphere. (E) Carbon utilization activity of rhizosphere bacterial community expressed as Average Well Color Development (AWCD). (F) Random Forest (RF) analysis to determine the importance of alpha diversity, carbon utilization activity, and structure of rhizosphere bacterial community in explaining tomato wilt disease. The linear regression showing the relationship between these factors and the wilt disease incidence. Symbols “C” and “S” represent the conducive soil and suppressive soil, while “C + R” and “S + R” represent the conducive soil and suppressive soil with R. solanacearum inoculation, respectively.
Adicionalmente realizan un perfil fisiológico a nivel comunitario de la fracción cultivable a travéz de la medición del consumo de carbono (AWCD), mostrando una ligera disminución entre la rizosfera de suelo propicio inoculado con Rasltonia C+R.
En la figura 5f hacen otro análisis de random forest e incluyen los valores del PCoA1 y 2 de la diversidad beta de la comunidad de las rizosferas del experimento de suelos S y C (supresor y propicio a la enfermedad respectivamente) y otras variables para intentar predecir qué factores explican la enfermedad del tomate, sin embargo esto puede traer consigo el problema de no saber qué aspecto de la diversidad beta es lo que está explicando una parte de este fenómeno.
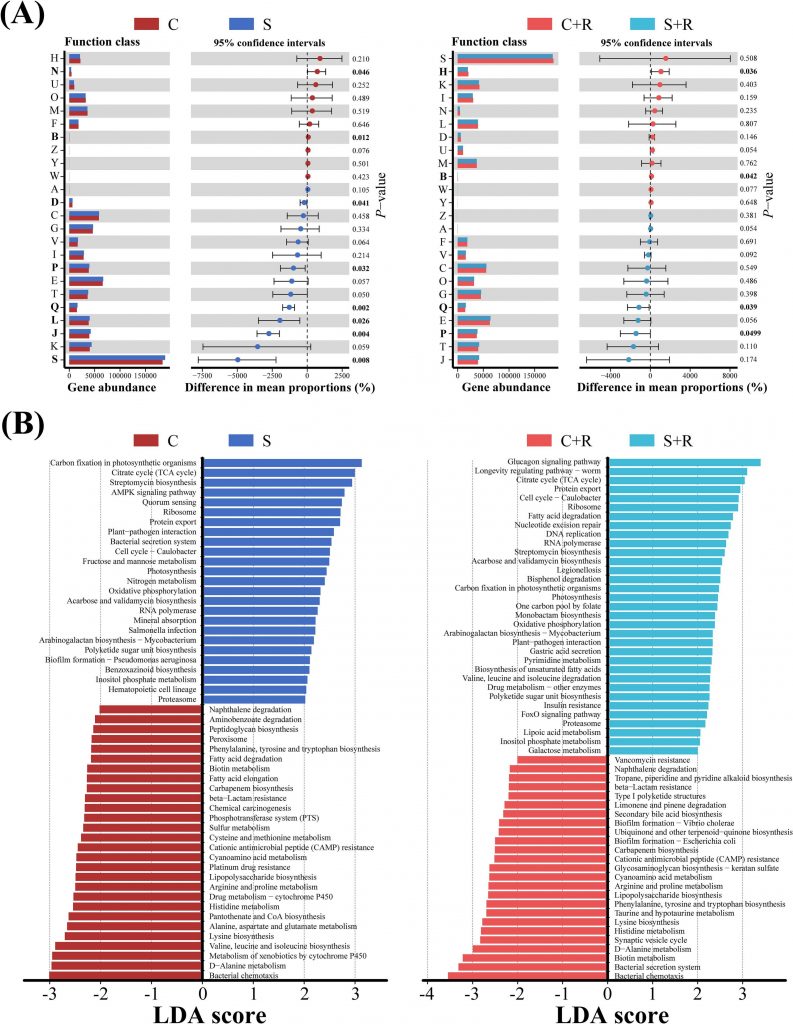
Fig. 6. Metagenome analysis of functional pathways differentially distributed in the rhizosphere soil of tomato plants grown in disease-conducive (C) and disease-suppressive (S) soils without and with R. solanacearum (R) (greenhouse trial 1). The analysis was performed using two different methods: a combination of the Statistical Analysis of Metagenomic Profiles (STAMP) and eggNOG database (A) or a combination of Lefse analysis (LDA score > 2, P < 0.05) and KEGG database (B). Symbols “C” and “S” represent the conducive soil and suppressive soil, while “C + R” and “S + R” represent the conducive soil and suppressive soil with R. solanacearum inoculation, respectively.
Referencia:
Peng, J., Hou, J., Liu, H., Mavrodi, D. V., Mavrodi, O. V., Sun, F., Shen, M., Wang, X., Dang, K., Yan, M., Liang, H., Dong, Y., & Li, J. (2025). Changes in the soil and rhizosphere microbiomes associated with bacterial wilt decline in the tomato monoculture field. Geoderma, 457, 117273. https://doi.org/10.1016/j.geoderma.2025.117273



