Estructura de la comunidad bacteriana del efluente de plantas de tratamiento de aguas residuales
Las plantas de tratamiento de aguas residuales (PTAR) reciben aguas residuales de distintas fuentes y son capaces de reducir la carga bacteriana de éstas. Sin embargo, los microorganismos contenidos en los efluentes de las PTARs pueden afectar al ecosistema en el que son depositados, propiciando la distribución de patógenos. En este trabajo se caracteriza la estructura y diversidad de las comunidades bacterianas presentes en el efluente final de dos PTARs (A y B) en Irlanda. Las PTARs se encontraban a menos de 100 km de distancia y se tomaron muestras del efluente durante tres días consecutivos en los meses de marzo y octubre en los años 2015 y 2016. De estas muestras se extrajo DNA y se amplificó y secuenció el gen 16S rRNA para poder analizar las comunidades bacterianas.
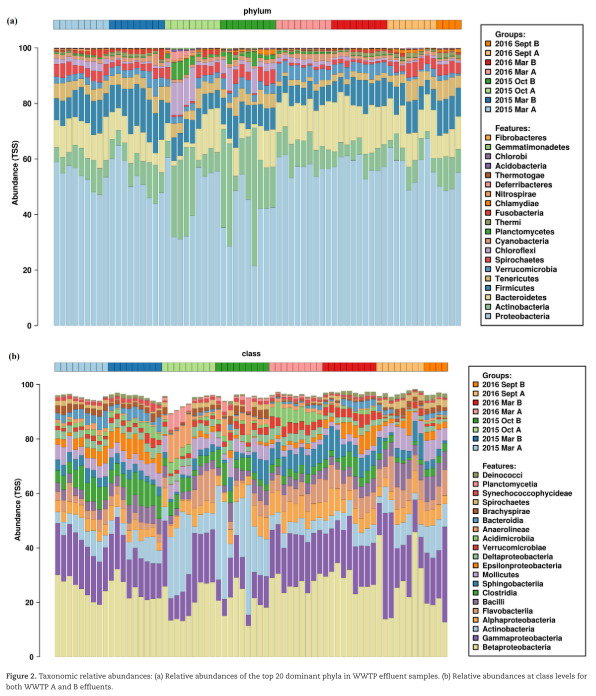
Los phyla que se detectaron en ambas PTARs fueron similares en todos los periodos de muestreo. Curiosamente, la composición de la comunidad bacteriana en las dos plantas varía en octubre de 2015, tanto a nivel de phyla como a nivel de clase. Esta variación no se relaciona con la temporada de lluvias de Irlanda (en octubre) aunque ya se ha reportado en otros estudios que puede deberse a cambios en temperatura. Para la PTAR A el índice de diversidad de Shannon fue mayor en primavera que en otoño, mientras que para la PTAR B fue mayor en otoño que en primavera. La riqueza (determinada por el estimador Chao1) fue más alta en primavera que en otoño para las dos PTARs. Los autores explican esto con la temperatura, ya que a temperaturas bajas (como las de primavera) se han reportado mayores valores de riqueza y una distribución más homogénea de los microorganismos presentes.
A nivel de género los autores muestran los 50 más abundantes en ambas PTARs y dentro de éstos se encuentran géneros reportados en el intestino humano (Flavobacterium, Acinetobacter, Bifidobacterium, Clostridium, Blautia, Bacteroides, Pseudomonas, Prevotella), en agua (Flavobacterium), suelo (Flavobacterium, Acinetobacter, Pseudomonas) e incluso algunos géneros relacionados con muestras fecales (Ruminococcus, Enterococcus, Dorea, Faecalibacterium). El 28% de estos 50 géneros son potenciales patógenos (ya que en estos géneros puede haber especies patógenas y no patógenas), de manera que las PTARs podrían considerarse posibles fuentes de distribución de patógenos al ambiente.
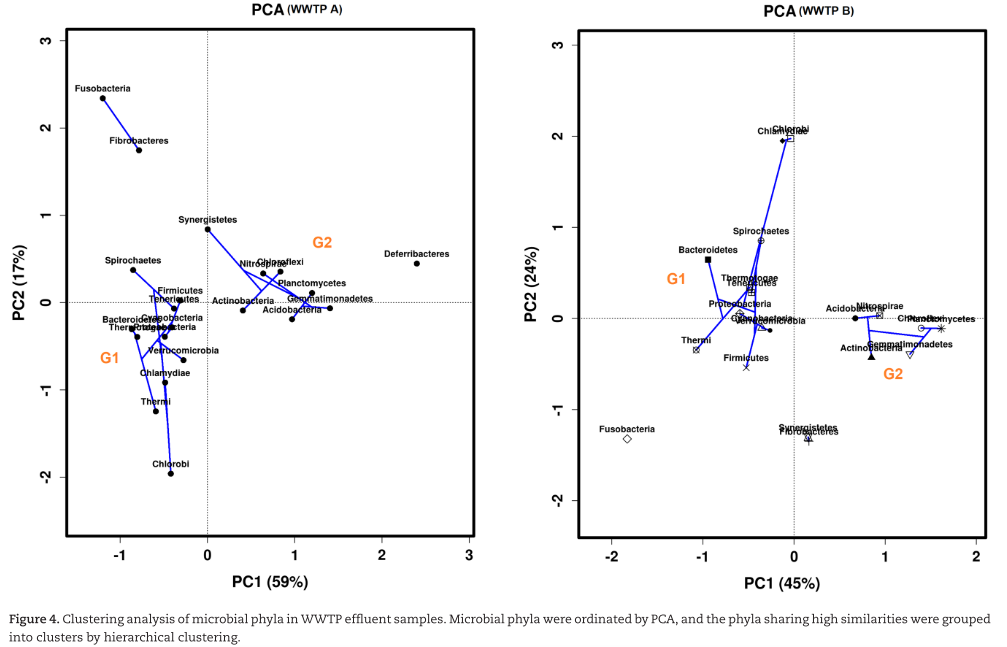
Al realizar un PCA se forman dos grupos que los autores denominan G1 y G2. Los phyla contenidos en el G1 de la PTAR A son los mismos contenidos en el G2 de la PTAR B. Esto se repite también para el G2 de ambas plantas. Esta agrupación parece haberse dado no por el año ni por ubicación de las PTARs, sino por la abundancia relativa de los phyla contenidos en cada grupo, ya que aquellos presentes en G1 son más abundantes que en el G2.
Referencia:
Do, T., Delaney, S., Walsh, F. (2019) 16S rRNA gene based bacterial community structure of wastewater treatment plant effluents. FEMS Microbiology Letters. 366.




