Maduración del microbioma supresor de patógenos durante el desarrollo de plantas
El microbioma de las plantas se encuentra relacionado a su estado de salud mediante efectos directos o indirectos. La supresión de patógenos es uno de los mecanismos por los cuales se mejora el estado de salud de las plantas. La mayoría de los estudios de microbiomas buscando a microorganismos supresores se han centrado en el aislamiento y caracterización de algunas cepas obtenidas durante algún estado de desarrollo de la planta, sin embargo se comprende poco sobre el efecto de la sucesión ecológica de las comunidades microbianas de rizosfera durante la maduración de las plantas y su impacto como comunidad en la protección contra patógenos.
En este trabajo, los autores caracterizan la composición y diversidad y abundancia del microbioma de plantas de jitomate en diferentes etapas de desarrollo (plántula, flor y fruto) mediante secuenciación masiva y qPCR del gen marcador rRNA 16S. Esto se complementó con el aislamiento de cepas en las que se probó la resistencia a estrés osmótico, nutricional y oxidativo, así como la producción y actividad de metabolitos secundarios inhibitorios para Ralstonia solanacearum y su antagonismo como una comunidad en contra del mismo patógeno.
La diversidad de OTUs disminuyó conforme el desarrollo de las plantas, mientras que la densidad (número de copias) bacteriana incrementó en las etapas de flor y fruto. Interesantemente, la composición global de las etapas tardías de desarrollo se diferencían a las de las plántulas y el suelo de origen, indicando la influencia del desarrollo de las plantas en el proceso de ensamblado del microbioma.
Más de la mitad de las cepas aisladas (193/331) fueron identificadas dentro de las bibliotecas de secuenciación masiva y su caracterización mostró que las representantes de comunidades de plantas florecientes o con fruto coinciden en tener una mayor diversidad de preferencia de fuentes de carbono y una mayor tolerancia a condiciones nutricionales pobres. De igual manera, las cepas de estas comunidades mostraron la mayor capacidad antagónica contra R. solanacearum en ensayos de toxicidad de metabolitos producidos y en condiciones de co-cultivo con la comunidad completa.
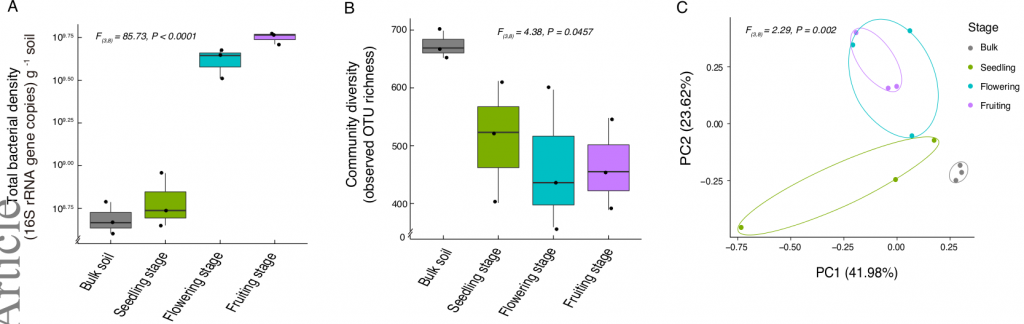
tomato growth stages, as measured with cultivation-independent DNA metabarcoding. Panel
A: 16S rRNA gene abundance per gram of rhizosphere soil as determined by qPCR. Panel B:
Metabarcoding-inferred microbiome diversity, measured as richness of observed OTUs with more
than ten reads per sample. Panel C: Community structure visualized by Principal component
analysis (PCA).
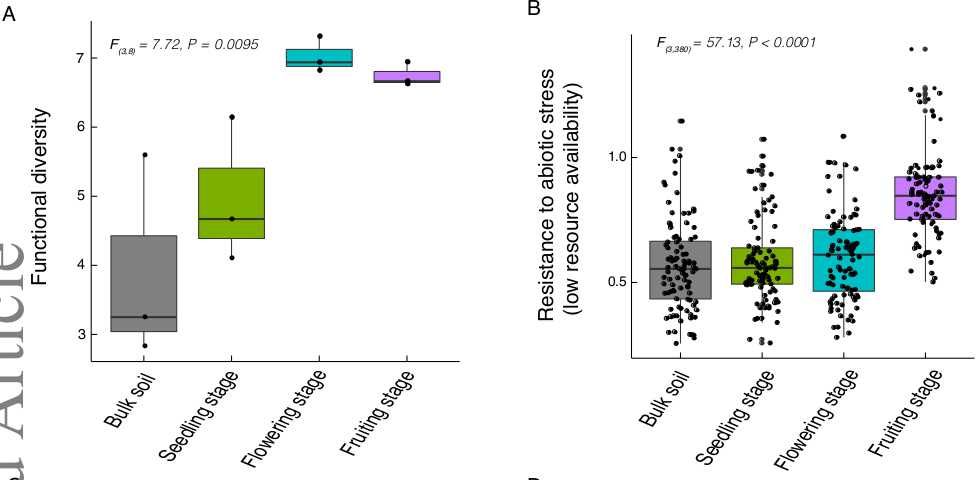
tomato plant development. Panel A: Functional diversity (FD) of cultivable bacteria isolated at
different plant growth stages. FD was defined on the base of resource use patterns of individual
isolates on representative plant-derived carbon sources, calculated independently for each plant
and time point (32 bacterial colonies per plant and time point). Panel B-D: Abiotic stress resistance
of bacteria isolated from tomato roots at different growth stages.
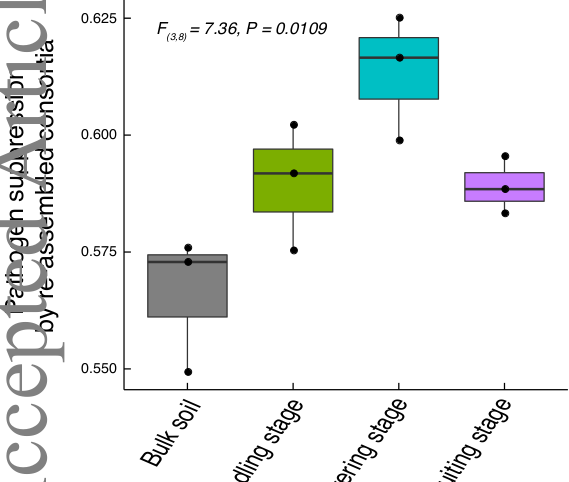
Consortia where re-assembled for each plant sample out of the 32 bacterial isolates isolated from
one plant sample and grown in liquid medium under invasion by the bacterial pathogen Ralstonia
solanacearum. Pathogen suppression was defined as the reduction of pathogen density at the end
of the experiment when the pathogen was co-cultured with the re-assembled consortia relative to
its density when grown in isolation. Each point represents one re-assembled consortium.
Los resultados del trabajo indican que las comunidades bacterianas de rizosfera tienen un proceso de sucesión ecológica dictado por el desarrollo de la planta, en donde la comunidad puede madurar hasta tener las mayores capacidades supresivas de un patógeno. Finalmente, la propuesta general en el texto de los autores resulta muy relevante debido a que se expone un punto sumamente importante, que es el enfocar este tipo de estudios a comunidades completas y tomar en cuenta la totalidad de interacciones que existen en este nicho
Hu, Jie, Zhong Wei, George A. Kowalchuk, Yangchun Xu, Qirong Shen, and Alexandre Jousset. «Rhizosphere microbiome functional diversity and pathogen invasion resistance build up during plant development.» Environmental Microbiology (2020).





