La red global de plásmidos en bacterias y arqueas
Los plásmidos son cromosomas accesorios del genoma bacteriano que pueden ser determinantes en el fenotipo bacteriano y ser transferidos horizontalmente. Estas transferencias de plásmidos ocurren primordialmente durante la conjugación bacteriana, en donde una bacteria forma un pili y transfiere información genética a otra bacteria compatible. Aunque este mecanismo de transferencia horizontal de genes (HGT, por sus siglas en inglés) es común entre bacterias de la misma especie, se sabe que pueden ocurrir conjugaciones entre bacterias de especies diferentes. Ademas, se sabe que los plásmidos son capaces de recombinar, generando linajes combinados de estos elementos genéticos.
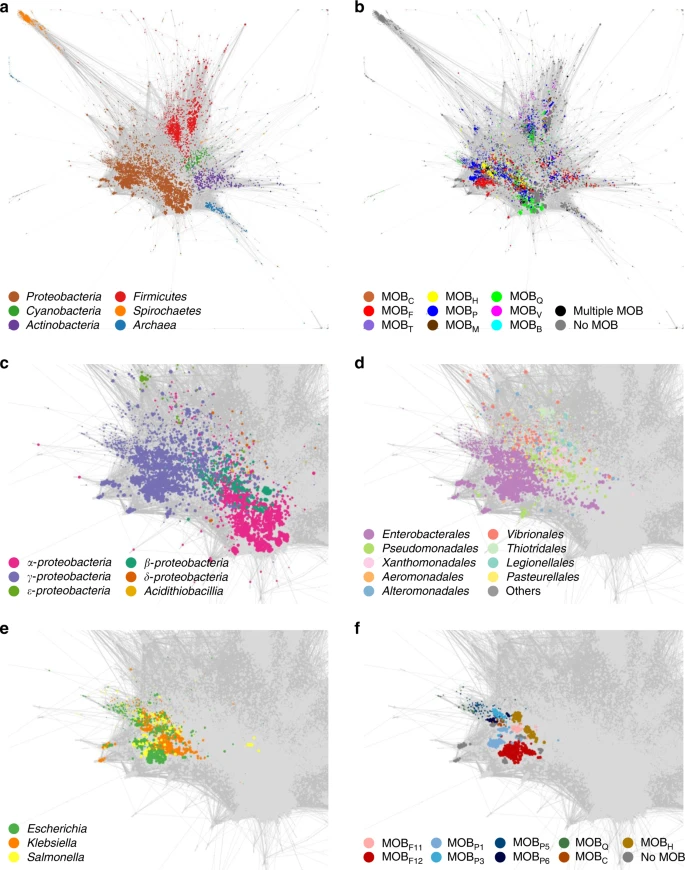
En este trabajo, Santiago Redondo-Salvo y compañía evaluaron si la evolución de los plásmidos conocidos se encuentra dirigida por las mismas fuerzas que la evolución de las especies bacterianas (como recombinación preferencial y cohesividad ecológica). Para ello, analizaron plásmidos de los genomas de bacterias y arqueas en la base de datos RefSeq para construir un mapa global del ‘plasmidoma’ por medio de redes en donde los nodos son plásmidos y los vértices son grupos de proteínas homólogas. Este mapa contiene varios ‘territorios’ que son grupos de plásmidos con proteínas homólogas que tienen una alta similitud. Los autores notaron que estos territorios se rigen por la afiliación filogenética de sus hospederos.
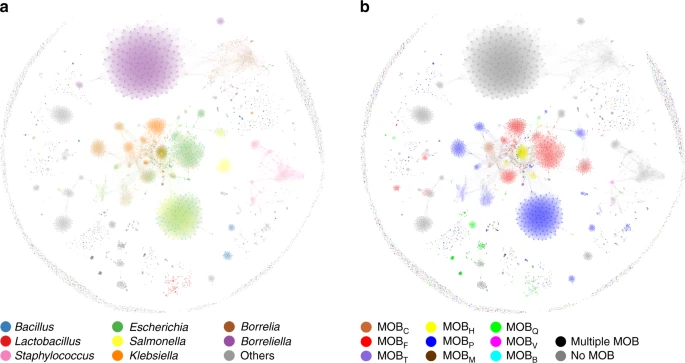
Similarity networks of the RefSeq84 prokaryotic plasmidome obtained using the ANIL50 algorithm as described in Methods.
Posteriormente, para evaluar la cohesividad genética de los plásmidos, deicidieron compararlos usando métricas con las que se determinan especies bacterianas, estas son la identidad nucleotídica promedio (ANI) y la fracción alineada (AF). Para visualizar estas relaciones, usaron un valor de corte de AF del 50% y formaron agrupamientos de por lo menos cuatro plásmidos que tuvieran una conectividad robusta y verificada con un modelo de bloque estocástico bayesiano. De esta forma, solamente en el orden Enterobacterales encontraron 83 agrupamientos que nombraron unidades taxonómicas plasmídicas (PTUs). Asimismo, observaron que los tipos funciones de movilización presentes en los plásmidos identificaban estas PTUs según las proteínas relaxasas que contienen. Estos resultados implican relaciones de similitud y la presencia de marcadores que se pueden usar para rastrear el origen común de los plásmidos, por lo cual los autores concluyeron su evolución es similar a las especies bacterianas.
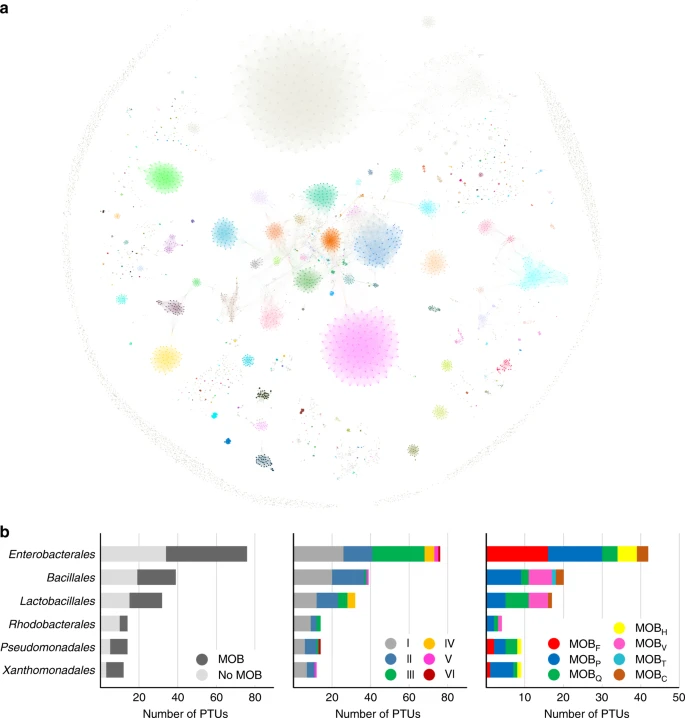
a A colormap of the PTUs identified by PID in the ANIL50 network of the entire bacterial plasmidome, retrieved from RefSeq84. Plasmids are colored by PTU membership. A comprehensive list of all PTUs and their members can be found in Supplementary Data 2. Source data are provided as a Source Data file. b Characteristics of PTUs found in the six more abundant orders.
Para poder trabajar con el plasmidoma entero, los autores diseñaron un algoritmo de identificación de plásmidos (PID) que verifica los agrupamientos eliminando centroides y probando la conectividad de los grupos. Tras analizar el plasmidoma completo, encontraron 276 PTUs, la mitad de ellos con relaxasas específicas. Las PTUs inmóbiles tuvieron una distribución de hospederos más limitada que las PTUs móbiles. Además el 81% de las unidades pertenecieron a dos clases solamente. Solo en seis órdenes bacterianos fue posible identificar más de 10 PTUs y estos presentaron el 75% de las unidades. De hecho, solo Enterobacterales, Bacillales, y Lactobacillales tuvieron el 50% de las PTUs identificadas. Los autores reconocen que este último resultado puede ser consecuencia del esfuerzo de muestreo en estos linajes bacterianos, ya que buena parte de su muestra estuvo compuesta por plásmidos observados una sola vez.
Santiago Redondo-Salvo y compañía concluyeron que a pesar de que la HGT de estos plásmidos tiene límites fijados por la filogenia, existen transferencias incluso al nivel de orden. Los autores tienen el mapa disponible en la siguiente liga: https://castillo.dicom.unican.es/PlasmidID/host-PTU/. Este trabajo nos ayuda a entender los patrones evolutivos de los plásmidos y nos inspira a seguir investigando las dinámicas de la evolución genómica en bacterias y arqueas.
Redondo-Salvo, S., Fernández-López, R., Ruiz, R. et al. Pathways for horizontal gene transfer in bacteria revealed by a global map of their plasmids. Nat Commun11, 3602 (2020). https://doi.org/10.1038/s41467-020-17278-2




