Un análisis sistemático de los grupos de genes biosintéticos del microbioma de mosquitos revela sideróforos antimaláricos que reducen la capacidad de reproducción de los mosquitos

La investigación del microbioma de insectos revela que una gran cantidad de compuestos metabólicos importantes para el desarrollo del mosquito provienen de las bacterias asociadas al microbioma. Cada uno de estas bacterias y cepas entre los grupos de bacterias ofrece un fenotipo o actividad que puede ser útil para el combate de diferentes plagas de insectos como lo pueden ser los mosquitos. Para estudiar esto los autores analizan la predicción de clusters de genes biosintéticos. Esta aproximación utilizada en diferentes organismos ahora tiene su alcance en el análisis de microbiomas de insectos.
Ya que hay reportes de diferentes substancias producidas por las bacterias en los mosquitos que afectan o benefician al mosquito en diferentes niveles. Los autores identifican la presencia de estos clusters buscando diferentes modos de afectar la transmisión de Plasmodium causante de la malaria.
En este trabajo identifican diferentes clusters biosintéticos de moléculas pequeñas de 33 bacterias asociadas al mosquito y donde identificaron más de 700 BGCs de los cuales los sideróforos son los más abundantes dentro del genoma de estas bacterias y están fuertemente asociadas a la intervención de la infección del mosquito por parte de Plasmodium.
Los 33 genomas fueron obtenidos de repositorios públicos y las bacterias corresponden a microorganismos aislados de diferentes especies de mosquitos de los géneros Aedes (dos especies) y Anopheles (cuatro especies). Todas estas bacterias asociadas al intestino de los mosquitos fueron seleccionadas por la presencia abundante dentro del metagenoma por estudios anteriores. Un árbol filogenético fue generado donde las bacterias más representadas son las proteobacterias, el segundo grupo más abundante fue el de las Elizabethkingia del filo Bacteroidetes. Esta última bacteria es de las más establecidas en el microbioma de los mosquitos después de la ingesta de sangre y azúcar.
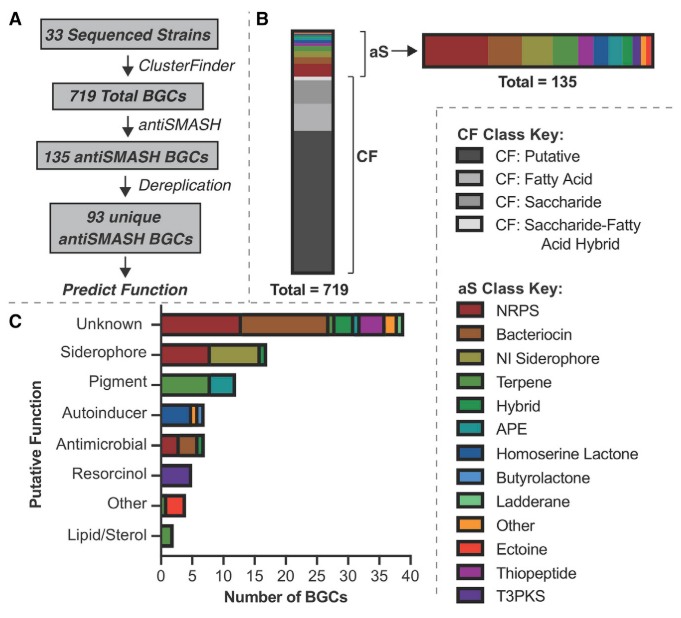
Cada bacteria fue analizada para identificar sus potenciales biosintéticos utilizando ClusterFinder. Un modelo de markov identifica los BGCs conocidos y desconocidos de una base de genomas. Los 719 BGCs obtenidos por CF fueron sometidos a antiSMASH para identificar los BGCs en clases. Eliminando redundancias 135 BSCs fueron identificados de varios tipo. Los más abundantes fueron los péptidos sintetasa no ribosomales (NRPS-IN) sideróforos, terpenos y bacteriocinas. Los clusters de NRPS (38/135) fueron los ams abundantes y se encontraron casi exclusivamente en las proteobacterias Chromobacterium, Serratia y Pseudomonas. Para asociar los metabolitos secundarios con los BGCs se asignaron funciones fisiológicas a los 135 y los clusters fueron comparados filogenéticamente con los productos caracterizados mas cercanos posibles con actividad biológica establecida.
group
Estableciendo funciones y metabolitos secundarios se eliminan las redundancias y se conservaron 93 BSCs anti-SMASH y fueron catalogadas con una de las siguientes funciones: desconocida, Sideroforo, pigmento, autoinductor, antimicrobiano, otros, resorcinol y lípidos/esterol.
Los sideróforos fueron los más abundantes 17 BGCs, 7 de ellos detectados en más de un organismos. Estos cumplen la función de secuestrar el Fe3+ y reducirlo para que pueda estar disponible para distintos procesos metabólicos. Estas funciones pueden estar estrechamente relacionadas con el establecimiento de Plasmodium en el intestino.

En este punto se realizó la identificación de sideróforos utilizando medios enriquecidos con Fe en cepas de bacterias asociadas a mosquitos utilizando cepas de Serratia, Pseudomonas y Enterobacter y detectando con espectrofotometría de masas. Entre lo encontrado detectaron BGC homólogos a desferrioxamina, bisucaberina, avaroferrina. Algunos de estos compuestos solo se habían detectado en bacterias entomopatógenas.
Para identificar el efecto en la sobrevivencia y establecimiento de Plasmodium en los mosquitos se sometieron mosquitos a diferentes sideróforos y se midieron diferentes factores de sobrevivencia.
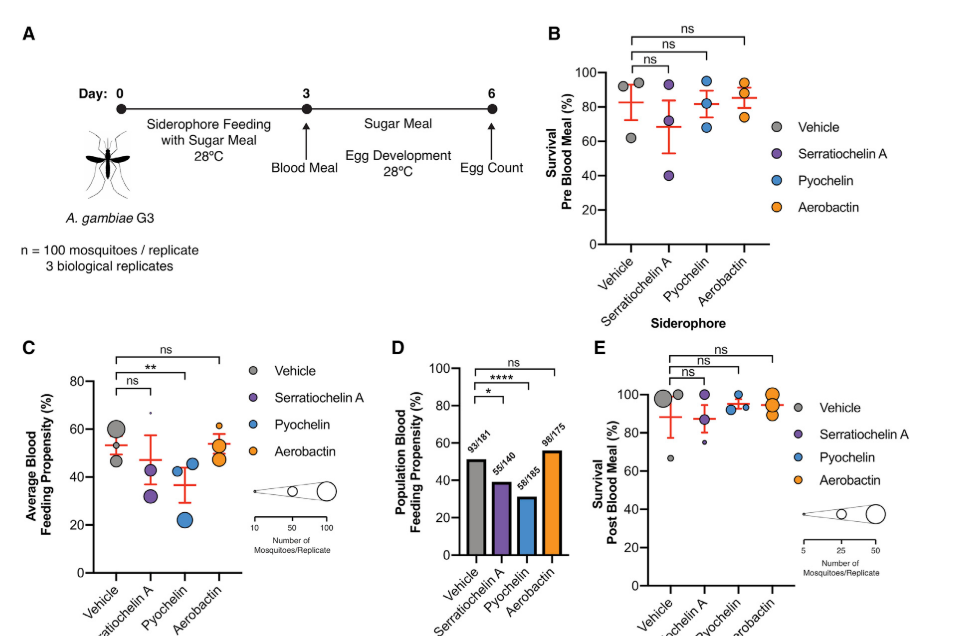
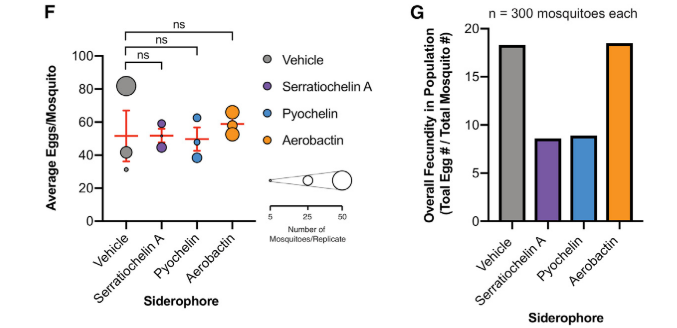
Los sideróforos como Serratiochelina A y Pyochelin tuvieron los niveles significativos más bajos de fecundidad de los huevos de mosquitos indicando el efecto del hierro en los diferentes procesos de reproducción de los mosquitos.
En cuanto a la evaluación de la actividad anti Plasmodium tres sideróforos disminuyeron la carga parasitaria en los mosquitos Serratiochelina, Pyochelina y Aerobactin. De esta manera los autores concluyen que los compuestos que reducen los niveles de hierro disponible, tienen potenciales efectos que reducen la transmisión de la malaria en los mosquitos

Ganley et al., A Systematic Analysis of Mosquito-Microbiome Biosynthetic Gene Clusters Reveals Antimalarial Side- rophores that Reduce Mosquito Reproduction Capacity, Cell Chemical Biology (2020), https://doi.org/10.1016/j.chembiol.2020.06.004




