Armando la secuencia completa de un cromosoma humano
A casi 20 años de la publicación del primer borrador del genoma humano, el ensamblado del genoma se encuentra fragmentado en cientos de secuencias continuas. Esto se debe a que el genoma humano tiene un alto contenido de secuencias repetitivas que generan confusión cuando se intenta armar la secuencia a partir de lecturas cortas de DNA. Aunque existen métodos de laboratorio para refinar los ensamblados y unir los fragmentos de cada cromosoma, la aparición de la tecnología de secuenciación por nanoporo ha permitido obtener lecturas de DNA lo suficientemente largas como para cubrir las repeticiones del genoma y resolver las confusiones del ensamblador. Con esto en mente, Karen Miga y compañía secuenciaron un genoma humano completamente homocigótico (CHM13, producido por una mola hidatiforme) con la plataforma MinION obteniendo 155 Gb y una profundidad de 50x que en teoría podría servir para armar el genoma efectivamente. La lectura más larga que recuperaron fue de 1.04 Mb y la mitad de las bases secuenciadas estuvieron contenidas en lecturas de por lo menos 70 kb.

Usaron el ensamblador Canu para lecturas largas agregando además lecturas de alta calidad secuenciadas con la plataforma PacBio. El primer ensamblado que generaron tuvo una mayor continuidad que el genoma de referencia actual (N50 de 75 Mb contra 56 Mb). Posteriormente, pulieron su armado con mapeo óptico y secuencias de nanoporo, PacBio e Ilumina, esta última tecnología en menor grado. Así obtuvieron un 90% de la secuencia de 6 cromosomas en dos contigs y diez cromosomas representados por dos scaffolds. Además de estar más completo que la referencia actual, su ensamblado tuvo una frecuencia balanceada para inserciones y deleciones a nivel estructural, una mayor concordancia con transcritos secuenciados y contiene 41 de las 46 secuencias teloméricas esperadas.
Debido a la alta continuidad en el ensamblado y a su importancia en la biología del ser humano, los autores decidieron terminar manualmente el armado del cromosoma X. Los saltos en las secuencias de este cromosoma se encontraban en el centrómero y en dos duplicaciones casi idénticas de más de 100 kb. Las duplicaciones fueron resueltas revisando manualmente lecturas largas independientes que abarcaban estas duplicaciones y posteriormente el armado fue validado por ddPCR. El centrómero representaba un desafío mayor dada su mayor longitud y su complicada combinación de repeticiones. Para armarlo, crearon un catálogo de las variantes estructurales y de un solo nucleótido dentro de la unidad DXZ1 y usaron las variantes como señalizaciones para ordenar las lecturas largas. Validaron su nuevo ensamblado con electroforesis de gel en campo pulsado, patrones de restricción, ddPCR y mapeos de las lecturas originales.
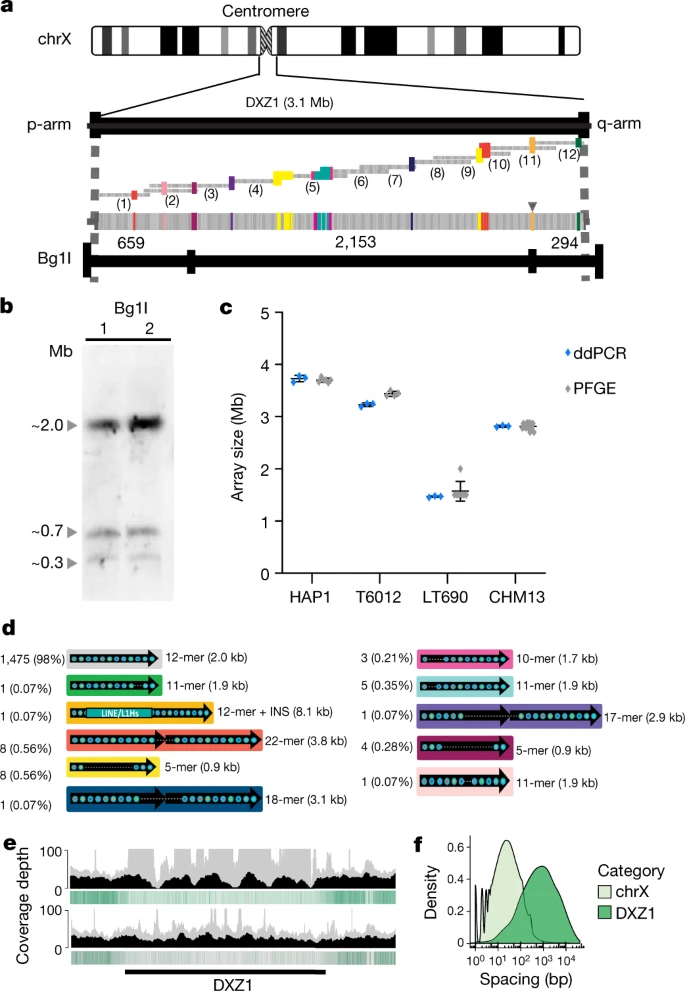
El ensamblado fue pulido con base en el llamado de bases con lecturas de las tres tecnologías ya mencionadas y al final se obtuvo una exactitud de 99.991% con respecto a BACs y del 99.995% con respecto a secuencias de Illumina. Además, se resolvieron más de un millón de pares de bases que representaban 29 gaps en el genoma de referencia. Posteriormente, Miga y compañía analizaron el perfil de metilación de este cromosoma, que puede ser leído por el nanoporo. Encontraron perfiles de metilación bajos en las regiones pseudoautosomales y perfiles variables en las repeticiones DXZ que fueron concordantes con estudios de genómica estructural. Los autores terminan comentando que analizar el resto del genoma humano es un todavía un desafío, sobre todo para los 5 cromosomas acrocéntricos. Este trabajo es un verdadero hito en el análisis genómico que servirá para convertir en conocimiento biológico las secuencias genómicas de los muchos organismos eucariontes.
Miga, K.H., Koren, S., Rhie, A. et al. Telomere-to-telomere assembly of a complete human X chromosome. Nature585, 79–84 (2020). https://doi.org/10.1038/s41586-020-2547-7





