El hallazgo de Candidatus Sukunaarchaeum mirabile, una Archaea con el genoma más pequeño hasta ahora descubierto
Uno de los grandes misterios de la biología moderna es entender cuál es el mínimo necesario para que algo sea considerado una célula viva. ¿Qué tan pequeña y simple puede ser una forma de vida celular antes de cruzar la línea hacia algo más parecido a un virus?. Un nuevo estudio ha arrojado una pieza clave para responder esta pregunta. Se trata del descubrimiento de una Archaea con el genoma más reducido jamás descrito para una célula autónoma.
Este microorganismo ha sido llamado Candidatus Sukunaarchaeum mirabile, y fue identificado en una célula marina de dinoflagelado (una especie de alga unicelular) que vive en simbiosis con otros microbios. Lo más llamativo de este hallazgo es que Sukunaarchaeum tiene un genoma de tan solo 238 mil pares de bases, lo que lo convierte en el genoma más pequeño conocido entre las Archaea, incluso más compacto que el del Nanoarchaeum equitans, que hasta ahora tenía el genoma más pequeño 490 mil pares de bases.
El hallazgo de Sukunaarchaeum es importante porque revela una forma de vida extrema, cuya única función aparente es replicarse, hasta donde sabemos. Esta archaea no posee rutas metabólicas comunes, no puede sintetizar aminoácidos, ni realizar funciones que tradicionalmente se consideran básicas para un organismo. Su genoma contiene principalmente genes relacionados con la replicación del DNA, la transcripción y la traducción, lo que constituye su «núcleo replicativo». Su funcionalidad se centra casi exclusivamente en la perpetuación de su información genética, lo cual lo acerca al territorio de los virus, que también dependen del metabolismo del huésped para replicarse. A diferencia de los virus, Sukunaarchaeum posee ribosomas y otros componentes esenciales para la síntesis de proteínas.
El genoma de Sukunaarchaeum fue detectado durante un estudio metagenómico realizado sobre un dinoflagelado marino, Citharistes regius. Harada y colaboradores aislaron una sola célula de este organismo y amplificaron el ADN de todos los microbios asociados a ella, encontraron el genoma completo de una cyanobacteria, dos proteobacterias, y un tercer genoma no identificado, circular, muy pequeño y con bajo contenido de GC (28.9%). Tras realizar un ensamblado híbrido (combinando lecturas cortas y largas de ADN), se encontró que este genoma contiene RNA ribosomal, 31 tRNAs, y varios genes esenciales típicos de archaea.
Mediante análisis filogenéticos se logró determinar que esta archaea forma una rama divergente, con secuencias de proteínas que evolucionan muy rápidamente. lo cual ha dificultado determinar con exactitud su relación filogenética, algunas técnicas lo ubican cerca del grupo Nanobdellati (formalmente DPANN Archaea), otros lo colocan cerca de los Halobacteriota perteneciente a Methanobacteriati (formalmente Euryarchaea). En cualquier caso, parece ser una rama nueva que podría representar un nuevo filo.
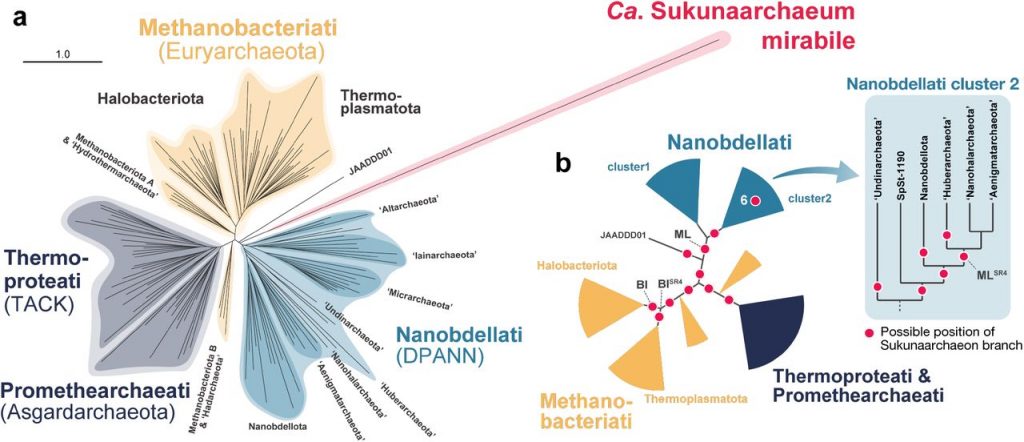
Figura que muestra las relaciones filogenéticas de Sukunaarchaeum con otros representantes del dominio Archaea, Figura tomada de Harada et al. (2025). A cellular entity retaining only its replicative core: Hidden archaeal lineage with an ultra-reduced genome. bioRxiv. https://doi.org/10.1101/2025.05.02.651781, bajo licencia CC-BY 4.0.
Además, utilizando datos del proyecto global Tara Oceans, Harada y colaboradores identificaron secuencias relacionadas con Sukunaarchaeum en distintas muestras marinas, lo que sugiere que en realidad pertenece a un grupo más amplio y diverso. Estas secuencias se encontraron principalmente en muestras de organismos marinos de tamaño de entre 5 a 20 micrómetros, lo que sugiere que Sukunaarchaeum y sus parientes viven asociados a células eucariotas pequeñas, probablemente como simbiontes o parásitos.
El análisis del contenido funcional del genoma de Sukunaarchaeum reveló que más del 70% de los genes con función conocida están dedicados al procesamiento de información genética, parece que no posee genes para metabolismo, rutas biosintéticas, ni producción de energía, con lo que Sukunaarchaeum parece depender completamente del entorno intracelular de su huésped.
A pesar de su genoma tan pequeño, Sukunaarchaeum contiene varios genes que codifican proteínas inusualmente grandes, algunas de más de 4,000 aminoácidos, y que representan hasta el 25% de su genoma. Análisis preliminares indican que estas proteínas podrían estar insertadas en la membrana celular y ser de función para interactuar con su huésped, tal vez facilitando la adhesión o el ingreso de nutrientes.
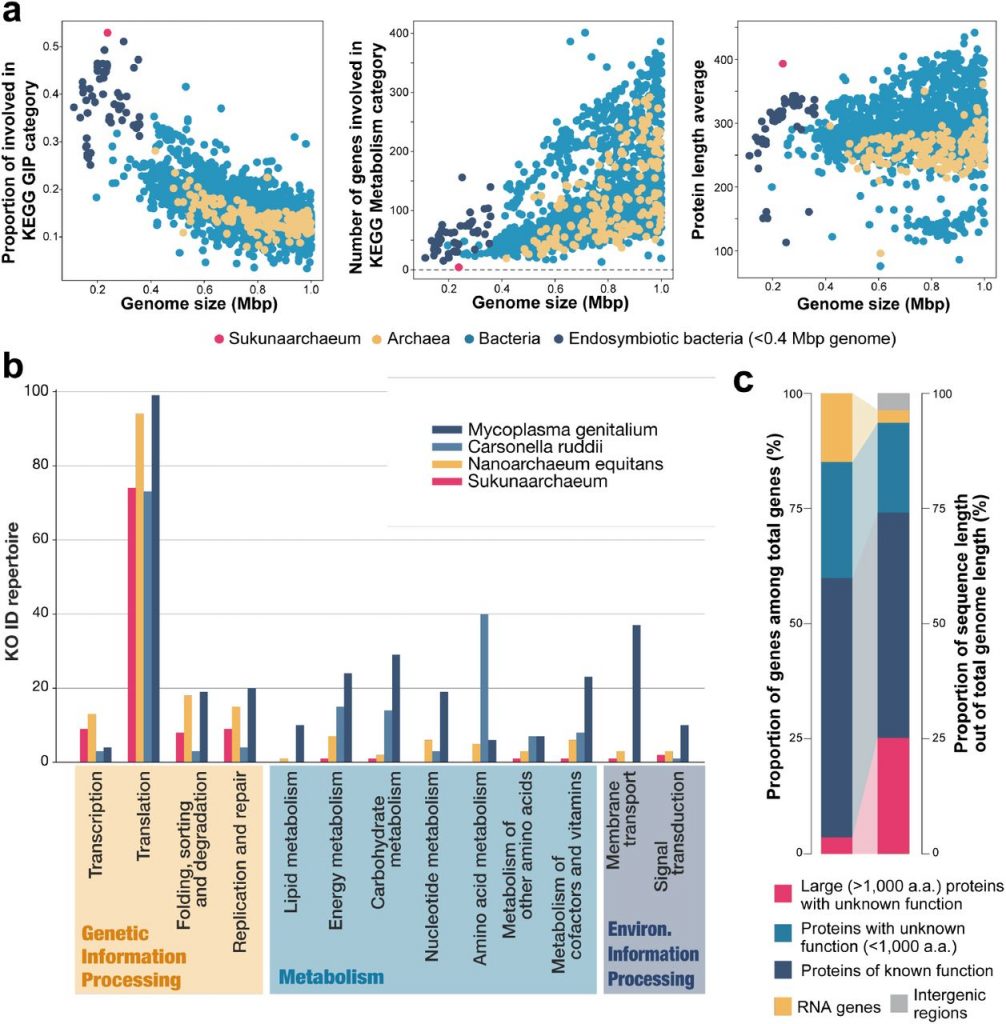
Figura que muestra la comparación genómica de Sukunaarchaeum mirabile con otros microorganismos de genoma reducido. Los gráficos muestran cómo casi todos sus genes están dedicados al procesamiento de información genética, la escasa presencia de genes metabólicos y que muchas de sus proteínas son sorprendentemente largas. Figura tomada de Harada et al. (2025). A cellular entity retaining only its replicative core: Hidden archaeal lineage with an ultra-reduced genome. bioRxiv. https://doi.org/10.1101/2025.05.02.651781, bajo licencia CC-BY 4.0.
El descubrimiento de Candidatus Sukunaarchaeum mirabile pretende redefinir los límites de lo que entendemos por vida celular, al mostrar que una célula puede sobrevivir sin metabolismo propio dependiendo por completo de otro organismo. Este descubrimiento nos muestra cuánto desconocemos aún del mundo microbiano, especialmente en contextos simbióticos.
Referencia:
Harada, R. et al. A Cellular Entity Retaining Only Its Replicative Core: Hidden Archaeal Lineage with an Ultra-Reduced Genome. bioRxiv. 2 May 2025, https://doi.org/10.1101/2025.05.02.651781.


