El atlas de proteomas de los tres dominios de la vida
El abaratamiento de los costos de secuenciación y el desarrollo de la genómica funcional ha permitido tener un inventario de las posibles funciones que tienen codificadas los organismos en sus genomas. Gran parte de estas funciones son levadas a cabo por proteínas y aunque se puedan predecir a partir de la secuencia genómica, eso no garantiza que en realidad se estén sintetizando en las células vivas. Esta incertidumbre aumenta cuando las proteínas detectadas por los algoritmos computacionales no tienen homólogos en bases de datos o no tienen alguna función conocida. Una forma de observar todas las proteínas de una célula o un tejido en un momento dado es la proteómica, que consiste en extraer todas estas moléculas de una muestra y sensarlas por medio de espectrometría de masas. En este trabajo, Johannes Müller y compañía llevaron a cabo esté análisis con 100 organismos diferentes, perteneciendo a los tres dominios de la vida: 19 arqueas, 49 bacterias y 32 eucariontes.
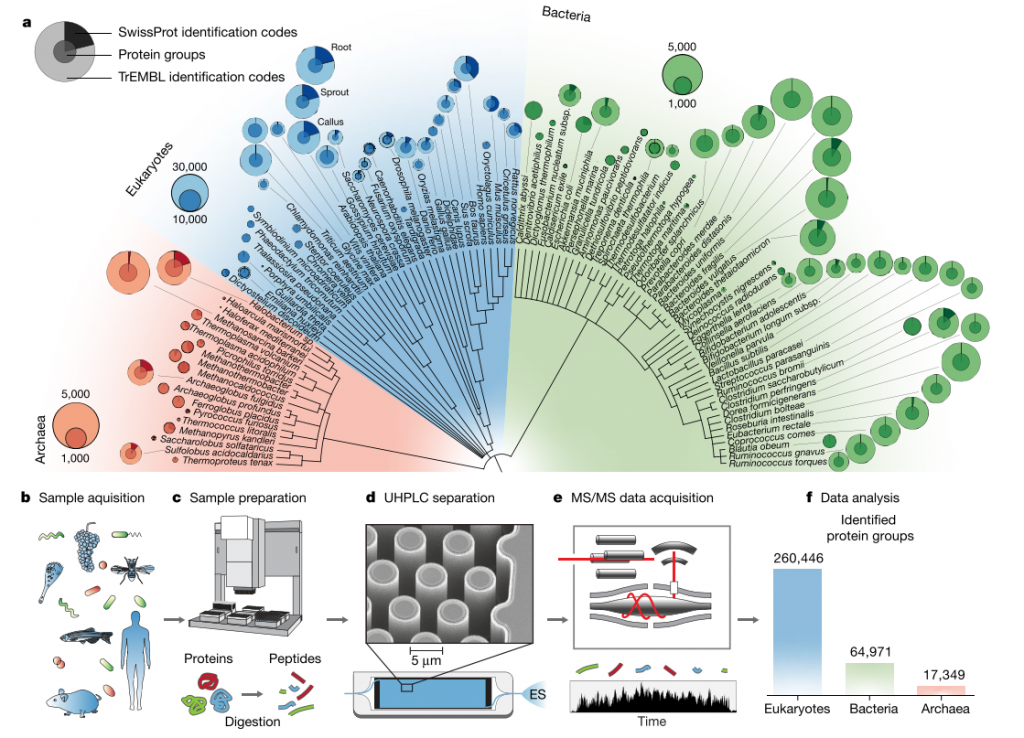
Para lograr esta proeza, aplicaron un modelo de aprendizaje profundo para predecir los tiempos de retención de las proteínas de sus organismos de estudio. Gracias a ello, pudieron recuperar una gran proporción de proteínas incluso en organismos sin datos de proteoma previos con un espectrómetro de masas de alto rendimiento. En total recuperaron 349,164 grupos de proteínas que cubrieron más de un millón de entradas de la base de datos de automatizada TrEMBL y un exceso de más de 800,000 proteínas sobre la base de datos Swiss-Prot (que contiene 559,634 proteínas). Esto aumenta de manera importante la cantidad de proteínas conocidas con evidencia experimental.

Para arqueas y bacterias, observaron casi la mitad de las proteínas predichas y en humanos encontraron 12 mil grupos de proteínas en 14 líneas celulares. El promedio del número de proteínas que representaban el 90% del contenido total de proteínas fue de 1,546 para eucariontes, 306 en bacterias y 262 en arqueas. Los términos de la ontología de genes más comunes fueron la traducción y plegamiento. Además, las funciones relacionadas con el ciclo de vida de las proteínas representó más del 10% de la masa total de proteínas observadas. Algunas anotaciones funcionales para las proteínas observadas fueron exclusivas para algunos grupos como la fotosíntesis en organismos foto autótrofos. Por otro lado, la fosforilación de proteínas tuvo una alta frecuencia en todos los organismos eucariontes. Finalmente, un 38.4% de las proteínas identificadas carece de anotación funcional, lo cual también representó casi el 23% de las 100 proteínas más abundantes.
Unas de las limitaciones reconocidas por los autores es la ausencia de condiciones distintas para obtener estos proteomas lo cual da como resultado un muestreo incompleto de las proteínas que sintetizan los organismos analizados. Este estudio es un gran hito en el análisis de proteínas ya que provee información de los tres dominios de la vida de forma estandarizada y tiene sus resultados disponibles en el portal proteomesoflife.org.
Müller, J.B., Geyer, P.E., Colaço, A.R. et al. The proteome landscape of the kingdoms of life. Nature (2020). https://doi.org/10.1038/s41586-020-2402-x




