Evaluando el efecto de normalizar cuentas por número de copias del 16S
El gen de rRNA 16S es el marcador molecular por excelencia en los estudios de ecología molecular para comunidades bacterianas. Aunque en teoría cada amplicón secuenciado representa a una célula de la comunidad, se sabe que muchos organismos pueden tener varias copias de este gen en su genoma e incluso se han detectado adquisiciones de copias del 16S por transferencia horizontal de genes. Anteriormente se ha propuesto hacer un inventario de los organismos que presentan varias copias de este gen y llevar a cavo una normalización por número de copias (GCN, por sus siglas en inglés), sin embargo no está claro si esta normalización en verdad aumenta la exactitud del análisis. Para comprobarlo, Robert Starke, Victor Satler y Daniel Kumazawa, decidieron tomar datos de comunidades bacterianas de las cuales se conocen las frecuencias relativas de sus miembros y ver si llevar a cabo la GCN hacía los resultados más apegados a los datos de referencia.

Los autores usaron la estrategia de las secuencias variantes de amplicones (ASVs) para analizar los datos y encontraron que hubo géneros de baja abundancia que no se encontraron o que no se identificaron. También observaron que las bacterias con varias copias del 16S tuvieron en promedio 6.6 copias del gen y mientras más ASVs sin identificar hubiera en la muestra, la desviación con respecto a las frecuencias de referencia aumentaba. Asimismo, los autores sugieren usar un promedio de 1.8 copias del gen 16S para estas secuencias sin identificar. La GCN fue efectiva en las comunidades Mock-21 y Mock-23, que eran las comunidades con la diversidad más baja. De igual forma, la comunidad Mock-14, que tuvo una alta diversidad, tenía miembros de los cuales se conoce a detalle el número de copias del 16S que contienen sus genomas.
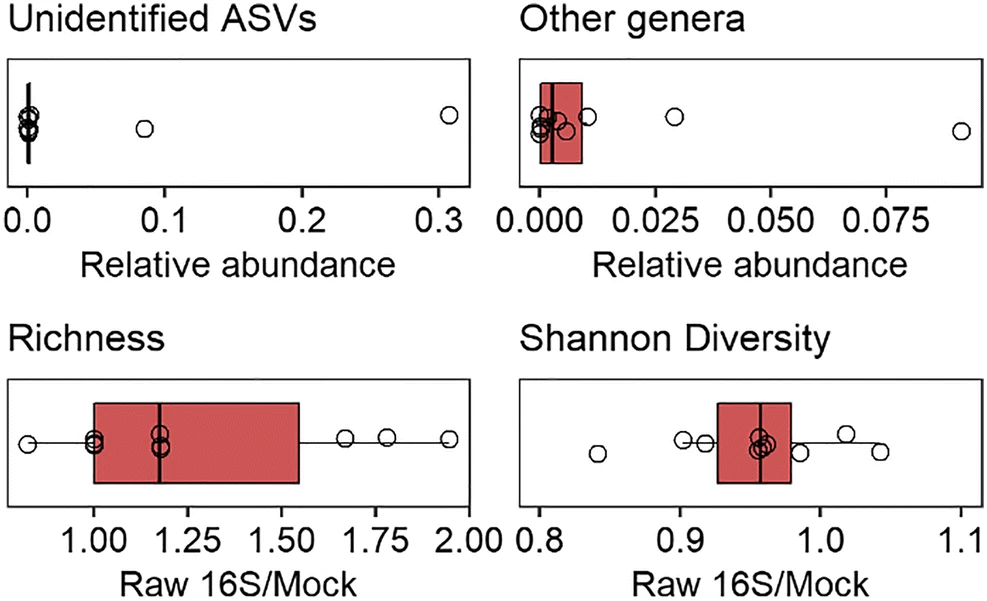
Starke, Satler y Kumazawa reconocen que los casos en los cuales la GCN fue efectiva, fueron artificiales ya que eran comunidades de muy baja diversidad y comunidades en donde el conocimiento previo de los miembros era atipicamente alto. Los autores sostienen que por el momento no vale la pena llevar a cabo la GCN, a menos que se cuente con un catálogo exhaustivo del número de copias del 16S de cada organismo. Este trabajo nos ayuda a entender las limitaciones de los análisis de microbiología ambiental.
Starke, R., Pylro, V.S. & Morais, D.K. 16S rRNA Gene Copy Number Normalization Does Not Provide More Reliable Conclusions in Metataxonomic Surveys. Microb Ecol (2020). https://doi.org/10.1007/s00248-020-01586-7


